Xác định trình tự gen mã hóa cytochrome B của hệ gen TY thể và mối quan hệ di truyền của loài cá heo ông sư (orcaella brevirostris gray, 1866) vùng biển Kiên giang, Việt Nam
Cá heo Ông Sư (Orcaella brevirostris Gray, 1866) còn có tên là cá heo
Irrawaddy, cá nược Minh Hải là một loài động vật có vú thuộc họ cá heo
Delphinidae. Đánh giá chung tình trạng của loài cá heo này theo Danh lục Đỏ IUCN,
cá heo Ông Sư được xếp vào bậc sẽ nguy cấp (VU - Vulnerable) và loài này được bảo
tồn ở Việt Nam theo Quyết định số 82/2008/QĐ-BNN của Bộ Nông nghiệp và Phát
triển Nông thôn.
Tuy nhiên, ở Việt Nam việc nghiên cứu về động vật biển nói chung trong đó
có loài cá heo Ông Sư vẫn còn rất ít các nghiên cứu. Bên cạnh đó kết quả nghiên cứu
về đặc điểm hình thái chưa đủ để định loại chính xác và đánh giá mối quan hệ di
truyền giữa quần thể cá heo Ông Sư Việt Nam với các quần thể cá heo Ông Sư khác.
Các tư liệu nghiên cứu di truyền loài cá heo này cũng không có nhiều [2]. Vì vậy,
nghiên cứu về đặc điểm hình thái kết hợp với phương pháp giám định phân tử trong
định loại và đánh giá mối quan hệ di truyền giữa quần thể cá heo Ông Sư Việt Nam
với các quần thể cá heo Ông Sư khác là cần thiết và có ý nghĩa khoa học.
Trong nghiên cứu này, nhóm tác giả sử dụng phương pháp sinh học phân tử
giải trình tự gen cytochrome b (cytb) và xác định mối quan hệ phả hệ của loài cá heo
Ông Sư tại vùng biển Kiên Giang, Việt nam

Trang 1

Trang 2
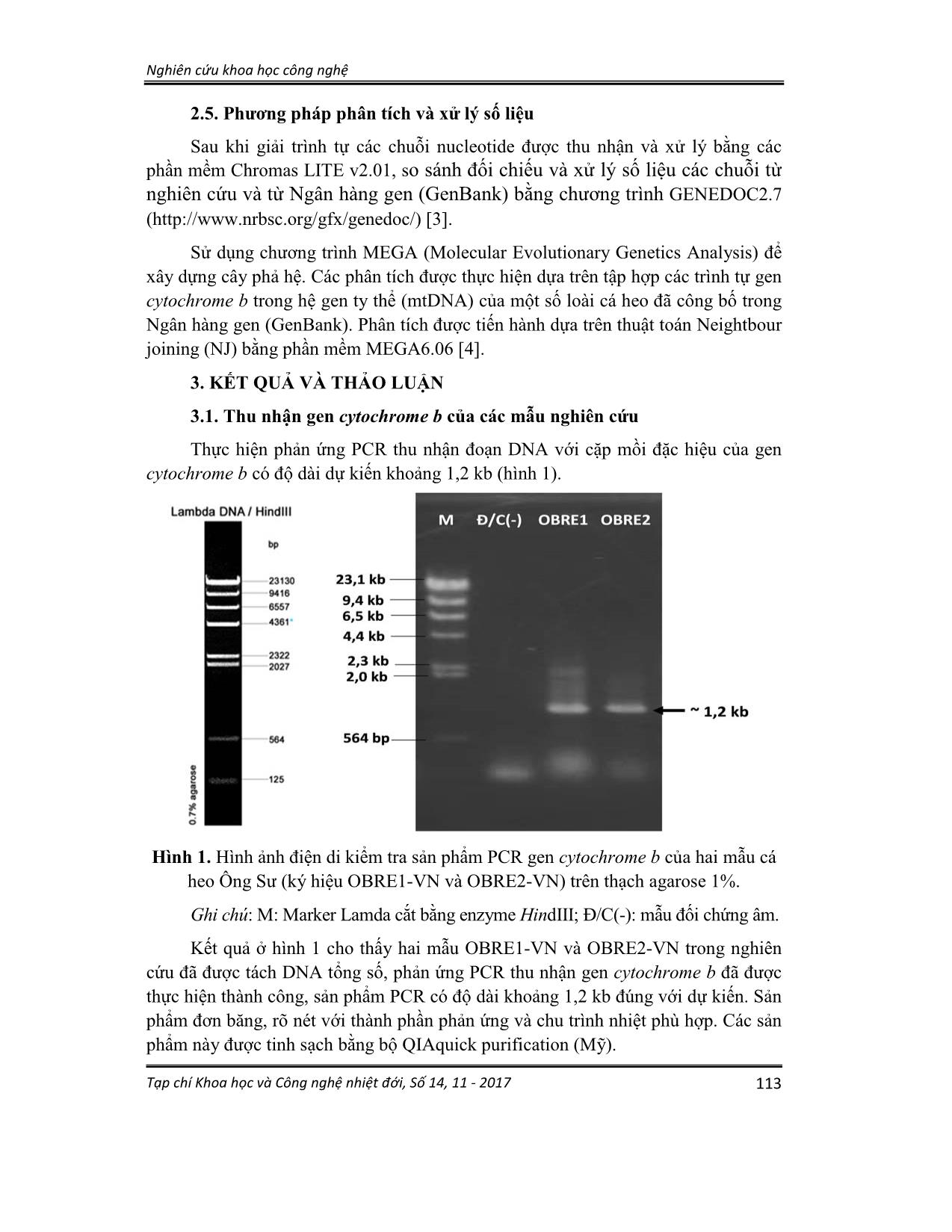
Trang 3

Trang 4
Trang 5

Trang 6

Trang 7

Trang 8
Tóm tắt nội dung tài liệu: Xác định trình tự gen mã hóa cytochrome B của hệ gen TY thể và mối quan hệ di truyền của loài cá heo ông sư (orcaella brevirostris gray, 1866) vùng biển Kiên giang, Việt Nam

Nghiên cứu khoa học công nghệ Tạp chí Khoa học và Công nghệ nhiệt đới, Số 14, 11 - 2017 111 XÁC ĐỊNH TRÌNH TỰ GEN MÃ HÓA Cytochrome b CỦA HỆ GEN TY THỂ VÀ MỐI QUAN HỆ DI TRUYỀN CỦA LOÀI CÁ HEO ÔNG SƯ (Orcaella brevirostris Gray, 1866) VÙNG BIỂN KIÊN GIANG, VIỆT NAM CÙ NGUYÊN ĐỊNH (1), NGUYỄN THỊ BÍCH NGA (2), NGUYỄN THỊ NGA (3), TRẦN LINH THƯỚC (4), BÙI LAI (5) 1. ĐẶT VẤN ĐỀ Cá heo Ông Sư (Orcaella brevirostris Gray, 1866) còn có tên là cá heo Irrawaddy, cá nược Minh Hải là một loài động vật có vú thuộc họ cá heo Delphinidae. Đánh giá chung tình trạng của loài cá heo này theo Danh lục Đỏ IUCN, cá heo Ông Sư được xếp vào bậc sẽ nguy cấp (VU - Vulnerable) và loài này được bảo tồn ở Việt Nam theo Quyết định số 82/2008/QĐ-BNN của Bộ Nông nghiệp và Phát triển Nông thôn. Tuy nhiên, ở Việt Nam việc nghiên cứu về động vật biển nói chung trong đó có loài cá heo Ông Sư vẫn còn rất ít các nghiên cứu. Bên cạnh đó kết quả nghiên cứu về đặc điểm hình thái chưa đủ để định loại chính xác và đánh giá mối quan hệ di truyền giữa quần thể cá heo Ông Sư Việt Nam với các quần thể cá heo Ông Sư khác. Các tư liệu nghiên cứu di truyền loài cá heo này cũng không có nhiều [2]. Vì vậy, nghiên cứu về đặc điểm hình thái kết hợp với phương pháp giám định phân tử trong định loại và đánh giá mối quan hệ di truyền giữa quần thể cá heo Ông Sư Việt Nam với các quần thể cá heo Ông Sư khác là cần thiết và có ý nghĩa khoa học. Trong nghiên cứu này, nhóm tác giả sử dụng phương pháp sinh học phân tử giải trình tự gen cytochrome b (cytb) và xác định mối quan hệ phả hệ của loài cá heo Ông Sư tại vùng biển Kiên Giang, Việt Nam. 2. ĐỐI TƯỢNG VÀ PHƯƠNG PHÁP NGHIÊN CỨU 2.1. Đối tượng nghiên cứu Cá heo Ông Sư được nuôi dưỡng tại Câu lạc bộ cá heo Vinpearl - Nha Trang. Đây là loài cá heo thu nhận tại vùng biển Kiên Giang - Việt Nam đã được thuần dưỡng. Ký hiệu 02 mẫu cá heo là OBRE1-VN và OBRE2-VN. Phương pháp lấy mẫu: Lấy mẫu máu toàn phần của cá cho vào ống có chất chống đông, bảo quản trong hộp xốp có đá để vận chuyển về tách DNA tổng số. 2.2. Phương pháp tách chiết DNA tổng số DNA tổng số được tách theo hướng dẫn của nhà sản xuất. Sử dụng bộ kit “Qiagen Genomic DNA Extraction Kit” của hãng QIAGEN Inc. (USA-Mỹ) để tách DNA tổng số. Nghiên cứu khoa học công nghệ Tạp chí Khoa học và Công nghệ nhiệt đới, Số 14, 11 - 2017 112 2.3. Phương pháp PCR thu nhận vùng gen đích Gen cytochrome b của hệ gen ty thể cá heo Ông Sư được lựa chọn để thu nhận gen bằng phương pháp PCR và giải trình tự, sau đó sử dụng chuỗi nucleotide của gen cytochrome b đã được giải trình tự để phân tích và so sánh với các chuỗi gen đã công bố trên Ngân hàng gen (Gen Bank). Mồi thực hiện PCR được thiết kế dựa trên trình tự bảo tồn của gen cytochrome b. Cặp mồi được sử dụng có trình tự trong bảng 1. Bảng 1. Trình tự các mồi thu nhận gen cytochrome b TT Tên mồi (primer) Trình tự mồi thiết kế 1 OBREF 5’-CAC ATC CCA TAG CAC CAC CC-3’ 2 OBRER 5’-AAT GGG GTC CTC CTT TTC GG-3’ Gen cytochrome b có kích thước khoảng 1.200 nucleotide (base pair - bp). Thực hiện phản ứng PCR với DNA tổng số thu nhận làm khuôn, sử dụng cặp mồi OBREF - OBRER để nhân đoạn gen đích bằng bộ kit AccuPower®ProFi Taq PCR Premix do hãng BIONEER (Hàn Quốc) cung cấp. Thành phần phản ứng PCR gồm: Khuôn DNA tổng số: 2 μl; Mồi xuôi (10pmol/μl): 2 μl; Mồi ngược (10pmol/μl): 2 μl; DMSO: 2 μl; Nước tinh khiết: 42 μl. Tổng thể tích là 50 μl. Chu trình nhiệt thực hiện PCR gồm: 1 chu kỳ 94oC - 5 phút; 35 chu kỳ 94oC - 30 giây; 52oC - 30 giây; 68oC - 7 phút; giai đoạn tổng hợp chuỗi: 72oC - 10 phút; Sản phẩm được bảo quản ở 4oC cho đến khi sử dụng. Điện di trên thạch agarose 1% để kiểm tra sản phẩm PCR và tiến hành tinh sạch sản phẩm PCR để loại bỏ các thành phần phụ không mong muốn chỉ giữ lại DNA tinh khiết. Tinh sạch sản phẩm bằng bộ QIAquick PCR purification kit (Qiagen Inc.). Sản phẩm PCR sau khi tinh sạch sẽ được dòng hóa vào vector tách dòng pCR2.1TOPO (hãng Invitrogen - Mỹ) để thu nhận DNA tái tổ hợp và gửi đọc trình tự tại công ty Macrogen (Hàn Quốc). 2.4. Phương pháp giải trình tự Cặp mồi dùng để giải trình tự DNA plasmid tái tổ hợp gồm: - Mồi xuôi (M13F) : 5’-GTAAAACGACGGCCAG-3’ hoặc - Mồi ngược (M13R) : 5’-CAGGAAACAGCATTGAC-3’ Chuỗi DNA sản phẩm được xác định trình tự theo chiều 5’→ 3’ và dùng thuốc nhuộm huỳnh quang (fluorescent dye) để đánh dấu, sử dụng thạch polyacrylamide và quét tia lazer dọc theo chuỗi để đọc trình tự, phân tích tự động bằng máy với các chương trình phần mềm tin - sinh học. Nghiên cứu khoa học công nghệ Tạp chí Khoa học và Công nghệ nhiệt đới, Số 14, 11 - 2017 113 2.5. Phương pháp phân tích và xử lý số liệu Sau khi giải trình tự các chuỗi nucleotide được thu nhận và xử lý bằng các phần mềm Chromas LITE v2.01, so sánh đối chiếu và xử lý số liệu các chuỗi từ nghiên cứu và từ Ngân hàng gen (GenBank) bằng chương trình GENEDOC2.7 ( [3]. Sử dụng chương trình MEGA (Molecular Evolutionary Genetics Analysis) để xây dựng cây phả hệ. Các phân tích được thực hiện dựa trên tập hợp các trình tự gen cytochrome b trong hệ gen ty thể (mtDNA) của một số loài cá heo đã công bố trong Ngân hàng gen (GenBank). Phân tích được tiến hành dựa trên thuật toán Neightbour joining (NJ) bằng phần mềm MEGA6.06 [4]. 3. KẾT QUẢ VÀ THẢO LUẬN 3.1. Thu nhận gen cytochrome b của các mẫu nghiên cứu Thực hiện phản ứng PCR thu nhận đoạn DNA với cặp mồi đặc hiệu của gen cytochrome b có độ dài dự kiến khoảng 1,2 kb (hình 1). Hình 1. Hình ảnh điện di kiểm tra sản phẩm PCR gen cytochrome b của hai mẫu cá heo Ông Sư (ký hiệu OBRE1-VN và OBRE2-VN) trên thạch agarose 1%. Ghi chú: M: Marker Lamda cắt bằng enzyme HindIII; Đ/C(-): mẫu đối chứng âm. Kết quả ở hình 1 cho thấy hai mẫu OBRE1-VN và OBRE2-VN trong nghiên cứu đã được tách DNA tổng số, phản ứng PCR thu nhận gen cytochrome b đã được thực hiện thành công, sản phẩm PCR có độ dài khoảng 1,2 k ... ...................................................................... : 168 Satt-z0002 : .................................................................................... : 168 Satt-z0002 : .................................................................................... : 167 Satt-z0011 : .................................................................................... : 168 Satt-z0011 : .................................................................................... : 168 Satt-z0012 : ..T................................................................................. : 168 Satt-z0016 : ...........................................................................-........ : 165 Satt-z0024 : .................................................................................... : 168 Satt-z0038 : .................................................................................... : 168 * 180 * 200 * 220 * 240 * OBRE1-VN-c : SVDKATLTRFFAFYFILPFIITALVIVHLLFLHETGSNNPTGIPSNMDMIPFHPYHTFKDILGALLLILVLLTLTLFTPDLLGD : 252 OBRE2-VN-c : .............H...................................................................... : 252 OBRE-KG-VN : .............H...................................................................... : 252 OBRE-Laos- : .............H...................................................................... : 252 OBRE-SWFSC : .............H...................................................................... : 252 Ohei02-DK- : .............H...........T.......................................................... : 252 Ohei06-DK- : .............H...........T.......................................................... : 252 Ohei08-DK- : .............H...........T..............................................M........... : 252 Ohei22-DK- : .............H...........T..............................................M........... : 252 Ohei28-DK- : .............H...........T..............................................M........... : 252 Oorc-72-US : .............H..........TA...............................I..T........T..A....A...... : 252 Oorc-96-US : .............H..........TA...............................I..T........T..A....A...... : 252 Oorc-97-US : .............H..........TA...............................I..T........T..A....A...... : 252 Oorc-111-U : .............H..........TA...............................I..T........T..A....A...... : 252 Oorc-129-U : .............H..........TA...............................I..T........T..A....A...... : 252 Oorc-ENAC2 : .............H..........TA...............................I..T........T..A....A...... : 252 Satt-z0000 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0002 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0002 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0002 : .............H..........AA.............................Y.I...........T..A........... : 251 Satt-z0011 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0011 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0012 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0016 : .............H..........AA.............................Y.I...........T..A........... : 249 Satt-z0024 : .............H..........AA.............................Y.I...........T..A........... : 252 Satt-z0038 : .............H..........AA.............................Y.I...........T..A........... : 252 260 * 280 * 300 * 320 * OBRE1-VN-c : PDNYTPANPLSTPAHIKPEWYFLFAYAILRSIPNKLGGVLALLLSILILIFIPMLQTSKQRSMMFRPFSQLLFWTLIADLLTLT : 336 OBRE2-VN-c : .................................................................................... : 336 OBRE-KG-VN : .................................................................................... : 336 OBRE-Laos- : .................................................................................... : 336 OBRE-SWFSC : .................................................................................... : 336 Ohei02-DK- : .................................................................................... : 336 Ohei06-DK- : .................................................................................... : 336 Ohei08-DK- : .................................................................................... : 336 Ohei22-DK- : .................................................................................... : 336 Nghiên cứu khoa học công nghệ Tạp chí Khoa học và Công nghệ nhiệt đới, Số 14, 11 - 2017 116 Ohei28-DK- : .................................................................................... : 336 Oorc-72-US : ...............................V.................................................... : 336 Oorc-96-US : ...............................V.................................................... : 336 Oorc-97-US : ...............................V.................................................... : 336 Oorc-111-U : ...............................V.................................................... : 336 Oorc-129-U : ...............................V.................................................... : 336 Oorc-ENAC2 : ...............................V.................................................... : 336 Satt-z0000 : ...............................................V.................................... : 336 Satt-z0002 : ...............................................V.................................... : 336 Satt-z0002 : ...............................................V.................................... : 336 Satt-z0002 : ...............................................V.................................... : 335 Satt-z0011 : ...............................................V.................................... : 336 Satt-z0011 : ...............................................V.................................... : 336 Satt-z0012 : ...............................................V.................................... : 336 Satt-z0016 : ...............................................V.................................... : 333 Satt-z0024 : ...............................................V.................................... : 336 Satt-z0038 : ...............................................V.................................... : 336 340 * 360 * 380 OBRE1-VN-c : WIGGQPVEHPYIIVGQLASILYFLLILVLMPTVSLIENKLLKW- : 379 OBRE2-VN-c : ...........................................- : 379 OBRE-KG-VN : ...........................................- : 379 OBRE-Laos- : ...S.......................................- : 379 OBRE-SWFSC : ...........................................- : 379 Ohei02-DK- : ...........................................- : 379 Ohei06-DK- : ...........................................- : 379 Ohei08-DK- : ...........................................- : 379 Ohei22-DK- : ...........................................- : 379 Ohei28-DK- : ...........................................- : 379 Oorc-72-US : ................................I..........- : 379 Oorc-96-US : ................................I..........- : 379 Oorc-97-US : ................................I..........- : 379 Oorc-111-U : ................................I..........- : 379 Oorc-129-U : ................................I..........- : 379 Oorc-ENAC2 : ................................I..........- : 379 Satt-z0000 : ................................AG.........- : 379 Satt-z0002 : ................................AG.........- : 379 Satt-z0002 : ................................AG.........- : 379 Satt-z0002 : ................................AG.........- : 378 Satt-z0011 : ................................AG.........- : 379 Satt-z0011 : ................................AG.........- : 379 Satt-z0012 : ................................AG.........- : 379 Satt-z0016 : ................................AG.....-...- : 375 Satt-z0024 : ................................AG.........- : 379 Satt-z0038 : ................................AG.........- : 379 Hình 3. Trình tự amino acid suy diễn gen cytochrome b của OBRE1-VN và OBRE2-VN so sánh với các chuỗi đăng ký trong Ngân hàng gen. Ký hiệu (.) là biểu thị các trình tự amino acid giống nhau. Các sai khác được thể hiện bằng các chữ cái. Kết quả ở hình 3 cho thấy chuỗi gen amino acid cytochrome b trong 2 mẫu OBRE1-VN và OBRE2-VN có sự tương đồng rất cao (99%) so với các chuỗi gen cytochrome b đã công bố trong Ngân hàng gen là OBRE-KG-VN (số đăng ký: JQ814471), mẫu của Indonesia OBRE-SWFSC-74697-ID (số đăng ký: JF289177) và mẫu của Lào là OBRE-Laos (số đăng ký: AF084062). Đặc biệt trong hai mẫu OBRE1-VN và OBRE2-VN phát hiện có sự sai khác tại vị trí amino acid Y182H (Y = Tyrosine; H = Histidine). Trong đó, Tyrorine là một acid amin không cần thiết có thể tổng hợp được trong cơ thể mà khi thiếu chúng thì cơ thể có thể bù trừ sự thiếu hụt đó nhờ các quá trình tổng hợp bên trong. Còn Histidine là một acid amin cần thiết không thể thay thế được vì chúng không thể tự tổng hợp trong cơ thể hoặc tổng hợp với tốc độ không thể đáp ứng được nhu cầu của cơ thể mà chúng phải được đưa vào đầy đủ trong đạm thức ăn. Về vấn đề này rất lý thú cần có thêm những nghiên cứu sâu hơn về bản chất của các amino acid và vai trò dinh dưỡng của chúng và với số lượng cỡ mẫu trong nghiên cứu cũng nhiều hơn. Trong nghiên cứu này, nhóm tác giả còn thu thập thêm một số chuỗi của loài Orcaella heinsohni (Ohei), Orcinus orca (Oorc) và Stenella attenuata (Satt) đăng ký trong GenBank thuộc họ cá heo để tham khảo trong so sánh và phân tích trình tự nucleotide và amino acid suy diễn. Kết quả xây dựng cây phả hệ dựa trên trình tự nucleotide chuỗi cytochrome b Nghiên cứu khoa học công nghệ Tạp chí Khoa học và Công nghệ nhiệt đới, Số 14, 11 - 2017 117 của 26 mẫu (bao gồm cả hai mẫu trong nghiên cứu) được trình bày ở hình 4. Kết quả cây phả hệ cho thấy hai mẫu OBRE1-VN và OBRE2-VN nằm trong cùng nhóm cá heo Ông Sư của Việt Nam (OBRE-KG-VN), Indonesia (OBRE-SWFSC-74697-ID) và Lào (OBRE-Laos). Như vậy, hai mẫu OBRE1-VN và OBRE2-VN đã được xác định chính xác thuộc loài cá heo Ông Sư (Orcaella brevirostris Gray, 1866). Hình 4. Cây phả hệ của OBRE1-VN và OBRE2-VN và các loài trong họ cá heo đã được đăng ký trong Ngân hàng gen dựa trên phân tích chuỗi nucleotide gen cytochrome b. Ký hiệu (♦) là các mẫu trong nghiên cứu này. 4. KẾT LUẬN 1. Sử dụng phương pháp sinh học phân tử so sánh, phân tích chuỗi nucleotide và amino acid suy diễn của gen cytochrome b. 2. Xây dựng cây phả hệ cho thấy hai mẫu OBRE1-VN và OBRE2-VN đã được xác định thuộc về loài cá heo Ông Sư có tên khoa học là Orcaella brevirostris Gray, 1866. 3. Các mẫu trong nghiên cứu có mối quan hệ phả hệ nguồn gốc gần với một số mẫu đã công bố trong Ngân hàng gen của Việt Nam, Indonesia và Lào. TÀI LIỆU THAM KHẢO 1. Bộ Nông nghiệp và Phát triển Nông thôn, Danh mục các loài thuỷ sinh quý hiếm có nguy cơ tuyệt chủng ở Việt Nam cần được bảo vệ, phục hồi và phát triển, Quyết định số 82/2008/QĐ-BNN, 2008. 2. LeDuc R.G., Perrin W.F., Dizon A.E., Phylogenetic relationships among the Delphinid cetaceans based on full cytochrome b sequences. Mar Mamm Sci 1999, 15:619-648. OBRE1-VN-cytB(1140bp) OBRE-KG-VN-JQ814471 OBRE2-VN-cytB(1140bp) OBRE-SWFSC-74697-ID-JF289177 OBRE-Laos-AF084062 Ohei02-JF339980 Ohei06-JF339981 Ohei08-JF339978 Ohei22-JF339979 Ohei28-JF339977 Oorc-96-KR180365 Oorc-97-KR180366 Oorc-72-KR180342 Oorc-111-KR180312 Oorc-129-KR180326 Oorc-ENAC2-Spain-HQ405752 Satt-z0011921-KX857288 Satt-z0012046-KX857298 Satt-z0016255-KX857317 Satt-z0024848-KX857334 Satt-z0000973-KX857266 Satt-z0038287-KX857341 Satt-z0002879-KX857280 Satt-z0002173-KX857273 Satt-z0002203-KX857279 Satt-z0011361-KX857281 100 89 100 92 100 75 100 100 76 0.005 Nghiên cứu khoa học công nghệ Tạp chí Khoa học và Công nghệ nhiệt đới, Số 14, 11 - 2017 118 3. Nicholas K.B., Nicholas H.B., GeneDoc: a tool for editing and annotating multiple sequence aglignments, Distributed by authors, 1997. 4. Tamura K., Stecher G., Peterson D., Filipski A., Kumar S., MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0, Mol Biol Evol, 2013, 30:2725-2729. 5. The IUCN Red List of Threatened Species. Version 2016.3. Truy cập 5/4/2017. SUMMARY DETERMINING THE MITOCHONDRIAL Cytochrome b GENE SEQUENCE AND THE HEREDITARY RELATIONSHIP OF IRRAWADDY DOLPHINS (Orcaella brevirostris Gray, 1866) IN KIEN GIANG WATERS, VIETNAM In this study, two samples of Irrawaddy dolphins (Orcaella brevirostris Gray, 1866) in Kien Giang waters, Vietnam were collected. The phylogenetic tree showed that Irrawaddy dolphins in Vietnam had a close genetic relationship to Irrawaddy dolphins in Indonesia and Laos which have been registered in the Gene Bank. This is the latest research in Vietnam about determining the mitochondrial cytochrome b gene sequence and the hereditary relationship of Irrawaddy dolphin (Orcaella brevirostris Gray, 1866) in Kien Giang waters. This results contributed to the conservative research and development of this valuable and rare marine mammal species. Từ khóa: Cá heo Ông Sư, phả hệ, phát sinh loài, ty thể, Orcaella brevirostris, cytochrome b, Irrawaddy dolphin, family tree, phylogenetic, mitochondria. Nhận bài ngày 24 tháng 8 năm 2017 Hoàn thiện ngày 12 tháng 10 năm 2017 (1) Chi nhánh Phía Nam, Trung tâm Nhiệt đới Việt - Nga (2) Viện Công nghệ Sinh học, Viện Hàn lâm KH&CN Việt Nam (3) Đại học Quốc tế Hồng Bàng (4) Đại học Khoa học Tự nhiên - ĐHQG.HCM (5) Viện Sinh học Nhiệt đới, Viện Hàn lâm KH&CN Việt Nam
File đính kèm:
 xac_dinh_trinh_tu_gen_ma_hoa_cytochrome_b_cua_he_gen_ty_the.pdf
xac_dinh_trinh_tu_gen_ma_hoa_cytochrome_b_cua_he_gen_ty_the.pdf

