Phát hiện và phân tách khối u não sử dụng phương pháp định biên và phát triển vùng
Trong lĩnh vực xử lý ảnh y khoa, phát hiện khối u não từ ảnh chụp cắt lớp vi tính (Computed tomography - CT) hoặc chụp cộng hưởng từ (Magnetic resonance - MRI) là một trong những mục tiêu ưu tiên hàng đầu. Trong bài báo này, chúng tôi mô tả một phương pháp mới kết hợp các thuật toán làm mịn, phát hiện biên Sobel, thành phần liên kết (Connected component), phát triển vùng (Region growing) để định vị và trích xuất các vùng tổn thương khác nhau một cách tự động, hiệu quả.

Trang 1

Trang 2

Trang 3

Trang 4
Trang 5
Trang 6
Trang 7
Trang 8

Trang 9

Trang 10
Tải về để xem bản đầy đủ
Bạn đang xem 10 trang mẫu của tài liệu "Phát hiện và phân tách khối u não sử dụng phương pháp định biên và phát triển vùng", để tải tài liệu gốc về máy hãy click vào nút Download ở trên
Tóm tắt nội dung tài liệu: Phát hiện và phân tách khối u não sử dụng phương pháp định biên và phát triển vùng

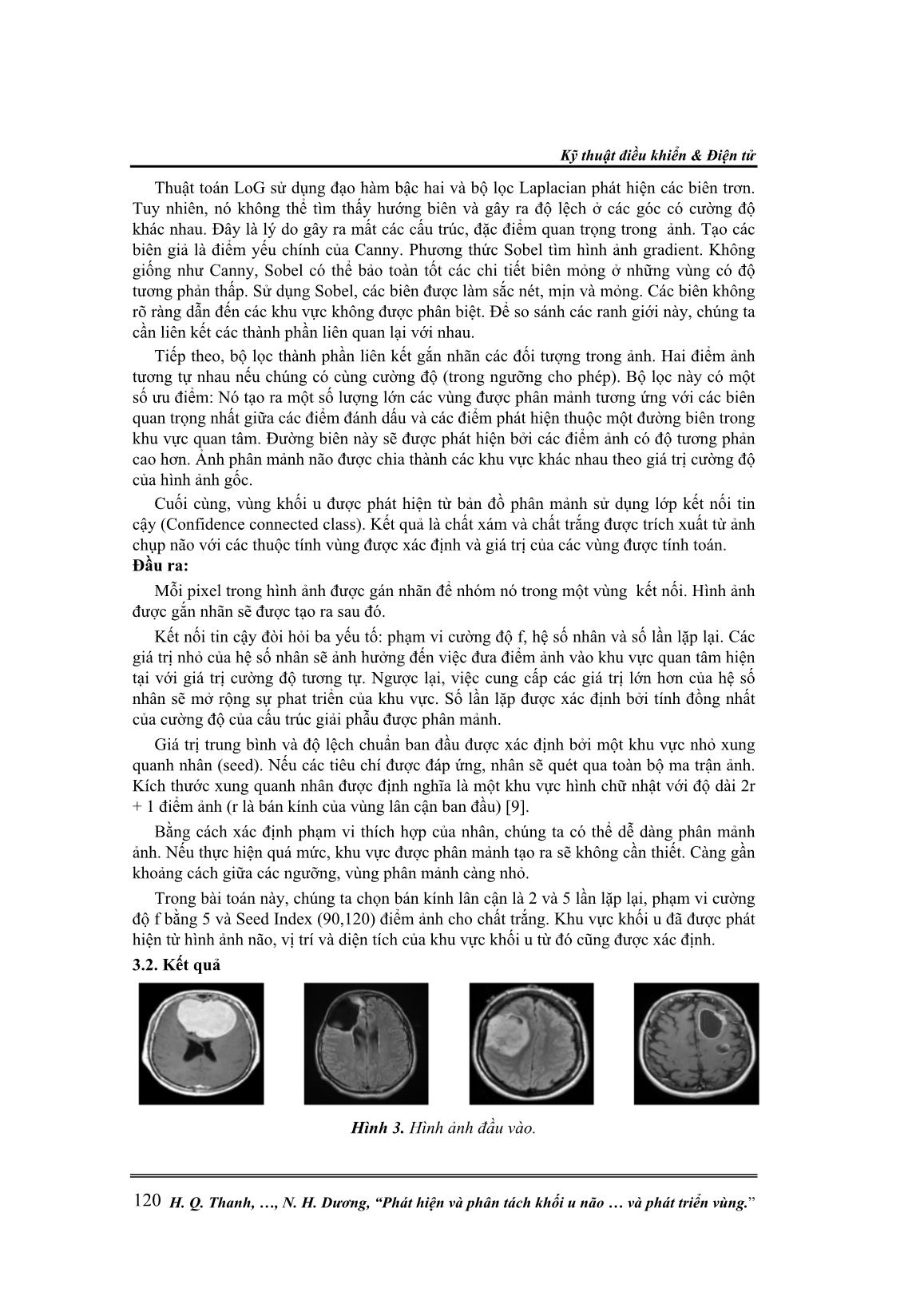
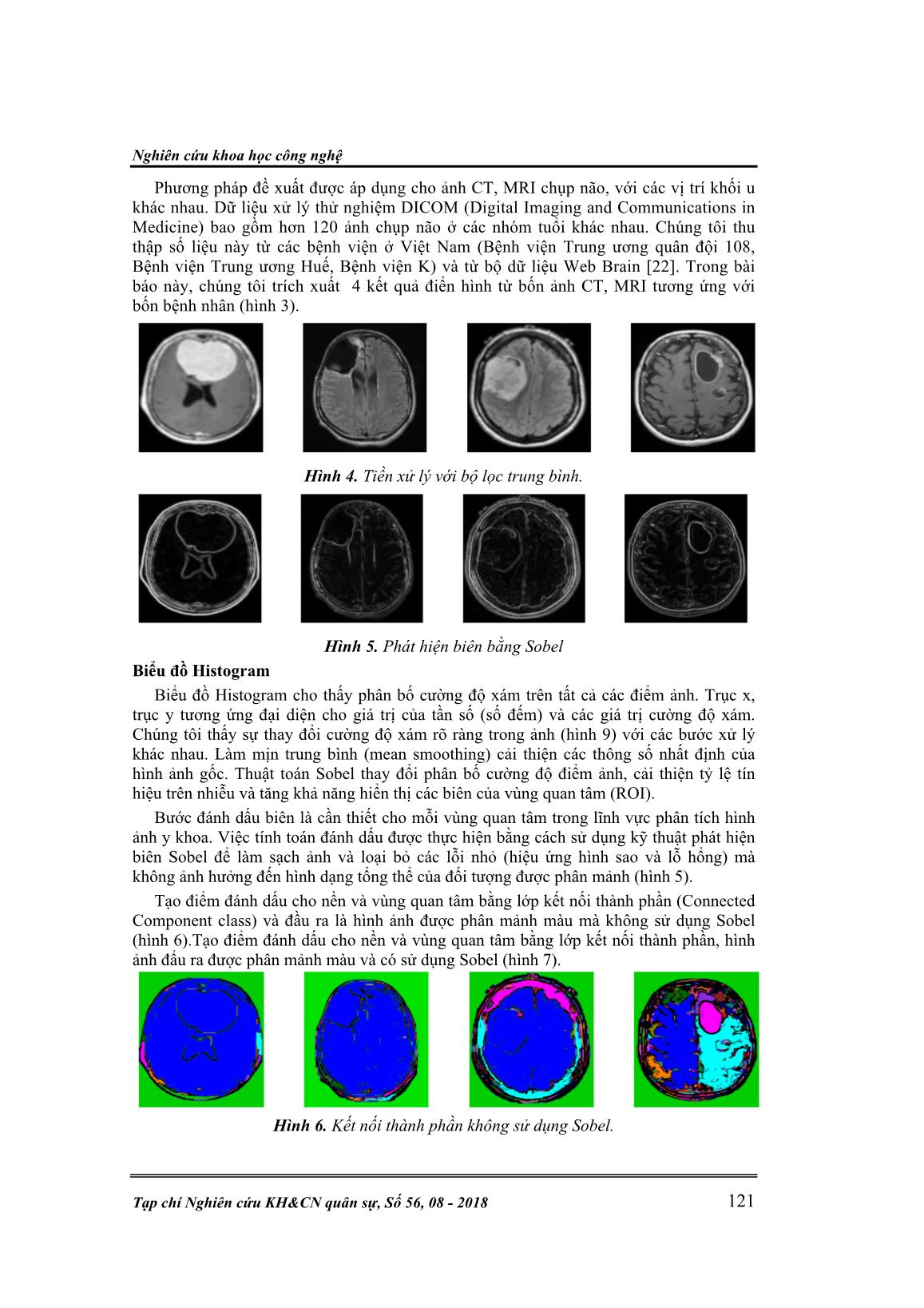
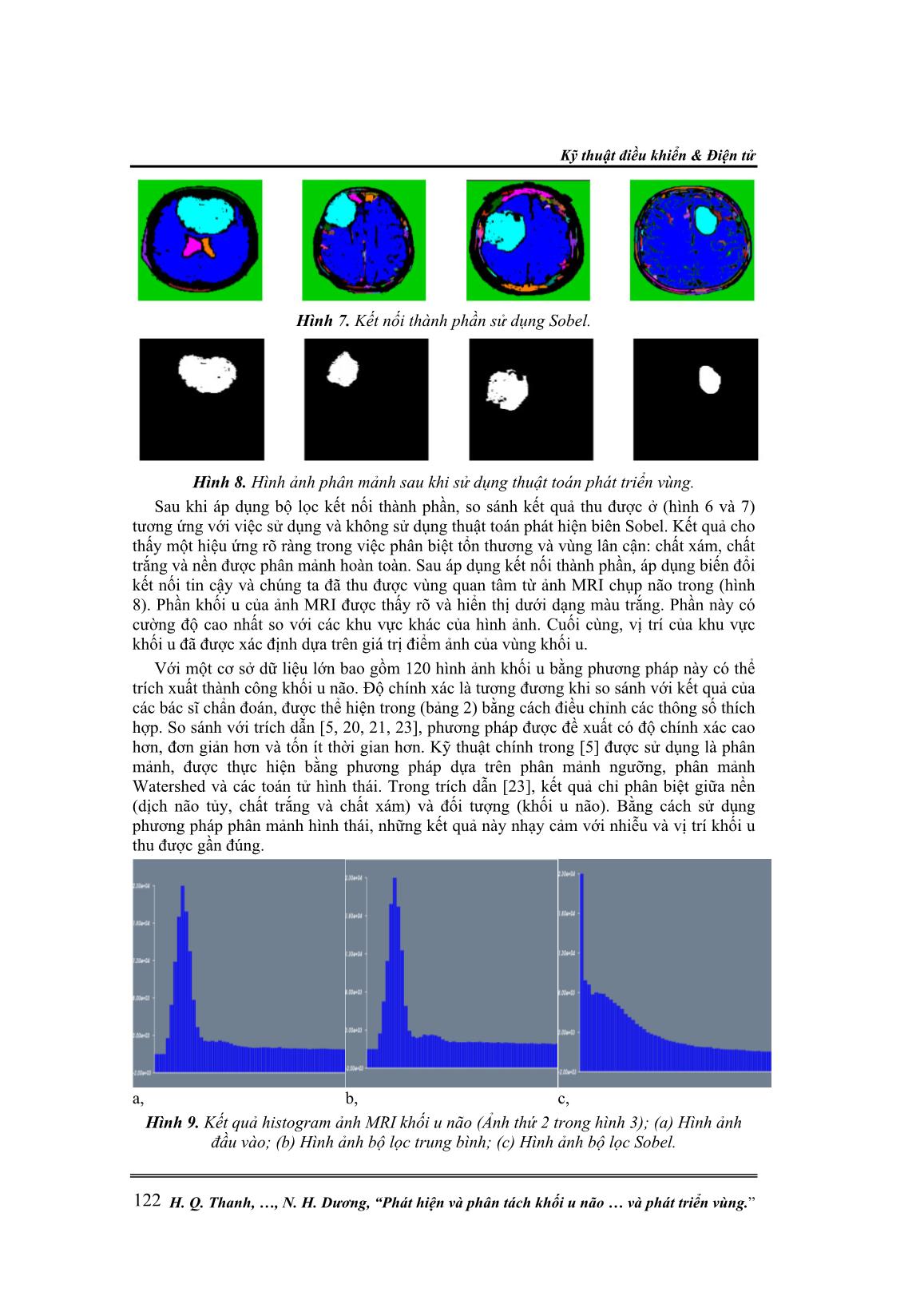
Nghiên cứu khoa học công nghệ Tạp chí Nghiên cứu KH&CN quân sự, Số 56, 08 - 2018 115 PHÁT HIỆN VÀ PHÂN TÁCH KHỐI U NÃO SỬ DỤNG PHƯƠNG PHÁP ĐỊNH BIÊN VÀ PHÁT TRIỂN VÙNG Hà Quang Thanh1*, Phan Việt Cương2, Hồ Thị Thảo2, Lê Tuấn Anh2, Nguyễn Hồng Hà2, Nguyễn Hải Dương3 Tóm tắt: Trong lĩnh vực xử lý ảnh y khoa, phát hiện khối u não từ ảnh chụp cắt lớp vi tính (Computed tomography - CT) hoặc chụp cộng hưởng từ (Magnetic resonance - MRI) là một trong những mục tiêu ưu tiên hàng đầu. Trong bài báo này, chúng tôi mô tả một phương pháp mới kết hợp các thuật toán làm mịn, phát hiện biên Sobel, thành phần liên kết (Connected component), phát triển vùng (Region growing) để định vị và trích xuất các vùng tổn thương khác nhau một cách tự động, hiệu quả. Hoạt động kết hợp này thực hiện một chu trình bao gồm các chức năng: kiểm tra quá trình ảnh chụp não, lọc ảnh, phân mảnh ảnh, xác định vị trí khối u và trích xuất vùng khối u. Thuật toán tính toán được thực hiện bằng cách sử dụng bộ công cụ Insight Toolkit (ITK) để xử lý ảnh đầu vào, Visualization Toolkit (VTK) để hiển thị và nền tảng xây dựng các ứng dụng Qt để xây dựng giao diện người dùng. Kết quả cho thấy phương pháp đề xuất đã phát hiện hiệu quả vùng khối u từ ảnh chụp não và tham chiếu với phantom. Từ đó phân tách phần bất thường khỏi phần mô lành bao quanh để thực hiện các xử lý hình ảnh nâng cao, tăng tỉ lệ thành công của liệu pháp điều trị bằng việc phát hiện sớm khối u cũng như giảm thời gian lập kế hoạch phẫu thuật, phác đồ điều trị. 1. GIỚI THIỆU Các khối u não được biết đến là một trong những nguyên nhân gây tử vong hàng đầu. Trong ảnh CT, MRI hình ảnh có sự chồng chéo giữa biên khối u và mô xung quanh hoặc các biên đó có thể bị che khuất bởi cấu trúc của hộp sọ dẫn đến giảm độ tương phản so với nền rất khó để phân biệt ranh giới giữa các mô bình thường và bất thường. Việc hiện thị rõ ràng và tách khối u mà không ảnh hưởng đến các mô xung quanh là một thách thức lớn đối với các bác sĩ [1]. Có rất nhiều cách tiếp cận được sử dụng trong nhiều nghiên cứu để phân biệt các đường biên sinh học của ảnh chụp não. Biji và cộng sự [2] đã đề xuất một kỹ thuật để phát hiện các khối u từ ảnh MRI sử dụng phân cụm mờ (fuzzy clustering) và ngưỡng sai số lỗi tối thiểu (minimum error thresholding). Phương pháp này khắc phục được vấn đề phân mảnh quá mức so với thuật toán watershed nhưng nhược điểm chính của phương pháp là tốn thời gian tính toán. Nagalkar V J và đồng nghiệp [3] đề xuất phân mảnh khối u não bằng cách sử dụng phương pháp tính toán mềm. Nhưng cách tiếp cận này tạo ra các biên giả trên hình ảnh. Bhattacharyya và đồng nghiệp [4] đã kết luận rằng một chuỗi thuật toán dựa trên thiết lập ngưỡng là một công cụ mạnh mẽ để phát hiện khối u não ảnh MRI. Phương pháp được đề xuất bởi Anam Mustaqeem, Ali Javed, Tehseen Fatima [5] yêu cầu thuật toán watershed cho phân mảnh. Nghiên cứu này đi vào phân tích giải phẫu não và triệu chứng, tổn thương do bệnh não gây ra. Nhược điểm duy nhất của đề xuất này là phân mảnh quá mức dẫn đến phát hiện kém các biên quan trọng và giảm độ tương phản thường thấy trong ảnh CT hoặc MRI não. M.C. Jobin Christ và R.M.S. Parvathi [6] đã giới thiệu phương pháp phát hiện khối u não bằng 2 cách : tích hợp các thuật toán phân cụm K- Means với Marker-controlled watershed và tích hợp thuật toán phân cụm Fuzzy C- Means và Marker-controlled watershed riêng biệt cho phân mảnh hình ảnh y khoa. Nhược điểm của phân nhóm K-means là đòi hỏi nhiều vòng lặp. Gibbs Kỹ thuật điều khiển & Điện tử H. Q. Thanh, , N. H. Dương, “Phát hiện và phân tách khối u não và phát triển vùng.” 116 và đồng nghiệp [7] sử dụng một quy trình phân mảnh trong đó các biên được phát hiện đầu tiên bằng cách sử dụng các toán tử hình thái và sau đó phân mảnh được thực hiện bằng cách sử dụng phương pháp phát triển vùng. M. Masroor Ahmed và cộng sự [8] đã đề xuất phương pháp phát hiện và trích xuất khối u não từ ảnh MRI bằng cách sử dụng phân cụm K-means. Phương pháp này rất hiệu quả, được chứng minh là tốn ít thời gian hơn và không bị mất dữ liệu khi nén hình ảnh. Hiện nay, thuật toán phân mảnh hiệu quả nhất thu được bằng cách kết hợp điều chỉnh các thuật toán khác nhau. Các tham số của các thuật toán này được điều chỉnh theo các thông số hình ảnh được sử dụng làm hình ảnh đầu vào và phụ thuộc vào các đăc điểm giải phẫu được phân mảnh [9]. Phân mảnh được thực hiện để hỗ trợ phát hiện, trích xuất và mô tả cấu trúc giải phẫu. Các kỹ thuật khác nhau của phân mảnh hình ảnh gồm: watershed, ngưỡng histogram, phân cụm K- means và phát triển vùng [10]. Phương pháp được đề xuất của nhóm nghiên cứu dựa trên cường độ của từng điểm ảnh. Cách tiếp cận này sẽ dễ dàng tách riêng biệt các khu vực bị ảnh hưởng. Ảnh cường độ (ảnh đa mức xám) được xem là ma trận dữ liệu, mỗi phần tử của ma trận tương ứng với một điểm ảnh và thể hiện cường độ của nó trong một phạm vi nhất định. Cường độ ảnh của mô não bình thường và vùng khối u được chia thành các nhóm khác nhau. Hình ảnh não bao gồm bốn vùng: chất trắng (white matter), chất xám (gray matter), dịch tủy não (cerebrospinal fluid) và nền (background). Các vùng này được gọi là các cụm với các điểm ảnh có cường độ khác nhau [11]. Phát hiện khối u là một quá trình xử lý mất nhiều thời gian. Do đó, nghiên cứu xác định vị trí, trích xuất, tính toán thể tích khối u tự động là cần thiết trong việc chẩn đoán và điều trị hiện nay. Trong bài báo này, chúng tôi sử dụng phương pháp phát triển vùng để phân mảnh, đánh dấu khu vực quan tâm (ROI) và nền trong hình ảnh đa mức xám. Quá trình này kết hợp các phương pháp cơ bản: làm mịn (smoothing), phát hiện biên và phân mảnh phát triển. Ở đây, phương pháp đề xuất bộ lọc làm mịn trung bình (mean smoothing) để giảm nhiễu trong ảnh CT, MRI. Thuật toán Sobel được sử dụng cho phát hiện biên ảnh, sử dụng thành phần kết nối để thiết lập ranh giới thích hợp giữa các vù ... ão với các thuộc tính vùng được xác định và giá trị của các vùng được tính toán. Đầu ra: Mỗi pixel trong hình ảnh được gán nhãn để nhóm nó trong một vùng kết nối. Hình ảnh được gắn nhãn sẽ được tạo ra sau đó. Kết nối tin cậy đòi hỏi ba yếu tố: phạm vi cường độ f, hệ số nhân và số lần lặp lại. Các giá trị nhỏ của hệ số nhân sẽ ảnh hưởng đến việc đưa điểm ảnh vào khu vực quan tâm hiện tại với giá trị cường độ tương tự. Ngược lại, việc cung cấp các giá trị lớn hơn của hệ số nhân sẽ mở rộng sự phat triển của khu vực. Số lần lặp được xác định bởi tính đồng nhất của cường độ của cấu trúc giải phẫu được phân mảnh. Giá trị trung bình và độ lệch chuẩn ban đầu được xác định bởi một khu vực nhỏ xung quanh nhân (seed). Nếu các tiêu chí được đáp ứng, nhân sẽ quét qua toàn bộ ma trận ảnh. Kích thước xung quanh nhân được định nghĩa là một khu vực hình chữ nhật với độ dài 2r + 1 điểm ảnh (r là bán kính của vùng lân cận ban đầu) [9]. Bằng cách xác định phạm vi thích hợp của nhân, chúng ta có thể dễ dàng phân mảnh ảnh. Nếu thực hiện quá mức, khu vực được phân mảnh tạo ra sẽ không cần thiết. Càng gần khoảng cách giữa các ngưỡng, vùng phân mảnh càng nhỏ. Trong bài toán này, chúng ta chọn bán kính lân cận là 2 và 5 lần lặp lại, phạm vi cường độ f bằng 5 và Seed Index (90,120) điểm ảnh cho chất trắng. Khu vực khối u đã được phát hiện từ hình ảnh não, vị trí và diện tích của khu vực khối u từ đó cũng được xác định. 3.2. Kết quả Hình 3. Hình ảnh đầu vào. Nghiên cứu khoa học công nghệ Tạp chí Nghiên cứu KH&CN quân sự, Số 56, 08 - 2018 121 Phương pháp đề xuất được áp dụng cho ảnh CT, MRI chụp não, với các vị trí khối u khác nhau. Dữ liệu xử lý thử nghiệm DICOM (Digital Imaging and Communications in Medicine) bao gồm hơn 120 ảnh chụp não ở các nhóm tuổi khác nhau. Chúng tôi thu thập số liệu này từ các bệnh viện ở Việt Nam (Bệnh viện Trung ương quân đội 108, Bệnh viện Trung ương Huế, Bệnh viện K) và từ bộ dữ liệu Web Brain [22]. Trong bài báo này, chúng tôi trích xuất 4 kết quả điển hình từ bốn ảnh CT, MRI tương ứng với bốn bệnh nhân (hình 3). Hình 4. Tiền xử lý với bộ lọc trung bình. Hình 5. Phát hiện biên bằng Sobel Biểu đồ Histogram Biểu đồ Histogram cho thấy phân bố cường độ xám trên tất cả các điểm ảnh. Trục x, trục y tương ứng đại diện cho giá trị của tần số (số đếm) và các giá trị cường độ xám. Chúng tôi thấy sự thay đổi cường độ xám rõ ràng trong ảnh (hình 9) với các bước xử lý khác nhau. Làm mịn trung bình (mean smoothing) cải thiện các thông số nhất định của hình ảnh gốc. Thuật toán Sobel thay đổi phân bố cường độ điểm ảnh, cải thiện tỷ lệ tín hiệu trên nhiễu và tăng khả năng hiển thị các biên của vùng quan tâm (ROI). Bước đánh dấu biên là cần thiết cho mỗi vùng quan tâm trong lĩnh vực phân tích hình ảnh y khoa. Việc tính toán đánh dấu được thực hiện bằng cách sử dụng kỹ thuật phát hiện biên Sobel để làm sạch ảnh và loại bỏ các lỗi nhỏ (hiệu ứng hình sao và lỗ hổng) mà không ảnh hưởng đến hình dạng tổng thể của đối tượng được phân mảnh (hình 5). Tạo điểm đánh dấu cho nền và vùng quan tâm bằng lớp kết nối thành phần (Connected Component class) và đầu ra là hình ảnh được phân mảnh màu mà không sử dụng Sobel (hình 6).Tạo điểm đánh dấu cho nền và vùng quan tâm bằng lớp kết nối thành phần, hình ảnh đẩu ra được phân mảnh màu và có sử dụng Sobel (hình 7). Hình 6. Kết nối thành phần không sử dụng Sobel. Kỹ thuật điều khiển & Điện tử H. Q. Thanh, , N. H. Dương, “Phát hiện và phân tách khối u não và phát triển vùng.” 122 Hình 7. Kết nối thành phần sử dụng Sobel. Hình 8. Hình ảnh phân mảnh sau khi sử dụng thuật toán phát triển vùng. Sau khi áp dụng bộ lọc kết nối thành phần, so sánh kết quả thu được ở (hình 6 và 7) tương ứng với việc sử dụng và không sử dụng thuật toán phát hiện biên Sobel. Kết quả cho thấy một hiệu ứng rõ ràng trong việc phân biệt tổn thương và vùng lân cận: chất xám, chất trắng và nền được phân mảnh hoàn toàn. Sau áp dụng kết nối thành phần, áp dụng biến đổi kết nối tin cậy và chúng ta đã thu được vùng quan tâm từ ảnh MRI chụp não trong (hình 8). Phần khối u của ảnh MRI được thấy rõ và hiển thị dưới dạng màu trắng. Phần này có cường độ cao nhất so với các khu vực khác của hình ảnh. Cuối cùng, vị trí của khu vực khối u đã được xác định dựa trên giá trị điểm ảnh của vùng khối u. Với một cơ sở dữ liệu lớn bao gồm 120 hình ảnh khối u bằng phương pháp này có thể trích xuất thành công khối u não. Độ chính xác là tương đương khi so sánh với kết quả của các bác sĩ chẩn đoán, được thể hiện trong (bảng 2) bằng cách điều chỉnh các thông số thích hợp. So sánh với trích dẫn [5, 20, 21, 23], phương pháp được đề xuất có độ chính xác cao hơn, đơn giản hơn và tốn ít thời gian hơn. Kỹ thuật chính trong [5] được sử dụng là phân mảnh, được thực hiện bằng phương pháp dựa trên phân mảnh ngưỡng, phân mảnh Watershed và các toán tử hình thái. Trong trích dẫn [23], kết quả chỉ phân biệt giữa nền (dịch não tủy, chất trắng và chất xám) và đối tượng (khối u não). Bằng cách sử dụng phương pháp phân mảnh hình thái, những kết quả này nhạy cảm với nhiễu và vị trí khối u thu được gần đúng. a, b, c, Hình 9. Kết quả histogram ảnh MRI khối u não (Ảnh thứ 2 trong hình 3); (a) Hình ảnh đầu vào; (b) Hình ảnh bộ lọc trung bình; (c) Hình ảnh bộ lọc Sobel. Nghiên cứu khoa học công nghệ Tạp chí Nghiên cứu KH&CN quân sự, Số 56, 08 - 2018 123 Bảng 2. Kết quả diện tích khối u được trích xuất. Ảnh Kích thước ban đầu (pixel) Diện tích theo pixel Diện tích khối u Độ chính xác (%) Ảnh 1 409 x 537 219633 32829 95.75 Ảnh 2 280 x 320 89600 12007 94.98 Ảnh 3 205 x 246 50430 10174 96.34 Ảnh 4 422 x 529 223238 8895 92.13 Chúng tôi tiến hành thử nghiệm phương pháp đề xuất trên phantom Gamex 463 được lấy từ bệnh viện ung bướu Kiên Giang. Kết quả tính toán diện tích của 2 vùng trên phantom lần lượt là 6475 pixel2 và 7230 pixel2, gần bằng với số liệu diện tích được ghi trên phantom là 6655 pixel2 và 7554 pixel2. Tỉ lệ chính xác tương ứng là 97.3% và 95.7%. a, b, c, d, Hình 10. (a) Ảnh gốc, (b) Ảnh thu được khi áp dụng Sobel, (c) Ảnh áp dụng kết nối thành phần, (d) Ảnh thu được sau khi áp dụng thuật toán phát triển vùng. Bằng cách sử dụng ITK với cấu hình hệ thống Inter(R) Core(TM)i5-4210U, CPU 1.7GHz, RAM 4GB, Windows 10 64 bit, thực hiện phương pháp tiếp cận được đề xuất trong [5, 20, 21, 23]. Kết quả chỉ ra rằng: Thời gian thực hiện phát triển vùng tốn ít hơn so với các phương pháp trích xuất khác được áp dụng cho số lượng dữ liệu 120 mẫu với kết quả đáng tin cậy, đồng thời thuật toán này cũng làm giảm độ phức tạp tính toán so với các thuật toán phân mảnh hình ảnh khác. 4. KẾT LUẬN Phương pháp đề xuất trên làm giảm sự phức tạp của một loạt các thuật toán phân mảnh hình ảnh. Tuy nhiên, không phải tất cả các kỹ thuật đều có tỷ lệ chính xác cao và cho kết quả mong muốn. Sử dụng ITK và VTK, chúng tôi đã chỉ ra sự kết hợp thuật phát hiện biên và phát triển vùng là phương pháp hiệu quả cho phân mảnh ảnh não. Do đó, hỗ trợ cho việc phân tích hình ảnh não trong các bệnh viện, phòng nghiên cứu. Phương pháp được đề xuất có khả năng phát hiện các khối u có kích thước nhỏ một cách nhanh chóng và chính xác khi so sánh với việc phát hiện thủ công bởi các chuyên gia y tế. Hiệu suất tính toán của nó cũng đã được chứng minh qua quá trình thử nghiệm trên phantom và với số lượng hình ảnh lớn (120 ảnh) trong thời gian thực hiện ít hơn so với phương pháp khác. TÀI LIỆU THAM KHẢO [1]. Grau V., Mewes A.U.J., Alcasiz M.: “Improved Watershed Transform for Medical Image Segmentation Using Prior Information”, IEEE Transactions On Medical Imaging, 2004, 23, pp. 447–458. [2]. Biji C.L., Selvathi D., Panicker A.: “Tumor detection in brain magnetic resonance images using modified thresholding techniques”, Communications in Computer and Information Sicence, 2011, 4, pp. 300–308. Kỹ thuật điều khiển & Điện tử H. Q. Thanh, , N. H. Dương, “Phát hiện và phân tách khối u não và phát triển vùng.” 124 [3]. Nagalkar V.J., Asole S.S: “Brain Tumor Detection using Digital Image Processing based on Soft Computing”, Journal of Signal and Image Processing, 2012, 3, pp. 102–105. [4]. Bhattacharyya D., Kim T.H.: “Brain tumor detection using MRI image analysis”, Communications in Computer and Information Science, 2011, 151, pp. 307–314. [5]. Anam M., Ali J., Tehseen F.: “An Efficient Brain Tumor Detection Algorithm using Watershed and Thresholding Based Segmentation”, International Journal Image, Graphics and Signal Processing, 2012, 10, pp. 34–39. [6]. Christ M.C.J., Parvathi R.M.S.: “Segmentation of Medical Image using Clustering and Watershed Algorithms”, American Journal of Applied Sciences, 2011, 8, pp. 1349–1352. [7]. Gibbs P., Buckley D.L., Blackband S.J., Horsman A.: “Tumour volume determination from MR images by morphological segmentation”, Physics in Medicine and Biology, 1996, 41, pp. 2437–2446. [8]. Ahmed M.M., Mohamad D.B.: “Segmentation of Brain MR Images for Tumor Extraction by Combining K-means Clustering and Perona-Malik Anisotropic Diffusion model”, International Journal of Image Processing, 2014, 2, pp. 27–34. [9]. Johnson H.J., McCormick M.M., Ibanez L.: “The ITK Software Guide”, https://itk.org/ItkSoftwareGuide.pdf, 2017, Chapter 4, pp. 79–82, pp. 343–358 [10]. S Jayaraman S., Esakkirajan S., Veerakumar T.: “Digital Image Processing”, (Tata McGraw Hill Education, 3 rd edn. 2010). [11]. Lim K.O., Pfefferbaum A.: “Segmentation of MR brain images into cerebrospinal fluid spaces, white and gray matter”, Journal of Computer Assisted Tomography, 1989,13, pp. 588–593. [12]. Anisha Bhatia: “Salt-and-pepper noise elimination in medical image based on median filter method”, International Journal of Electrical, Electronics and Data Communication, 2013,1, pp. 22–24. [13]. Nagabhushana S.: “Computer Vision and Image Processing”, New Age International Publishers, 2005, pp. 73–202. [14]. Hua X.C., Yi C.J, Bin X.W.: “Medical image denoising by generalised Gaussian mixture modelling with edge information”, IET Image Processing, 2014, 8, pp. 464–476. [15]. Xu.P, Miao Q., Shi C., Zhang J., Yang M.: “General method for edge detection based on the shear transform”, IET Image Processing, 2012, 6, pp. 839–853. [16]. Sharma D., Jindal G.: “Computer Aided Diagnosis System for Detection of Lung Cancer in CT Scan Images”, International Journal of Computer and Electrical Engineering, 2011, 3, pp. 714–718. [17]. Mehena J.: “Medical Image edge detection based on mathematical morphology”, International Journal of Computer and communication technology, 2013, 2, pp. 49–53. [18]. Jiang A., Chuang L., Lu Y., Fahn C.S.: “Mathematical-morphology-based edge detectors for detection of thin edges in low-contrast regions”, IET Image Process, 2007, 1, pp. 269–277. [19]. Kumar S.N, Fred A.L., Ajay K.H, Varghese S., Manikandan M.: “Medical image Edge detection using Gauss Gradient operator”, Journal of Pharmaceutical Sciences and Research, 2017, 9, pp. 695–704. [20]. Mehena J., Adhikary M.C.: “Brain Tumor Segmentation and Extraction of MR Images Based on Improved Watershed Transform”, IOSR Journal of Computer Engineering, 2015, 17, pp. 01–05. Nghiên cứu khoa học công nghệ Tạp chí Nghiên cứu KH&CN quân sự, Số 56, 08 - 2018 125 [21]. Malathi R., Kamal N.A.R.: “Brain Tumor Detection and Identification Using K- Means Clustering Technique”, Proceedings of the UGC Sponsored National Conference on Advanced Networking and Applications, 2015, pp. 14–18. [22]. “Brain web:”, [23]. Mohane S., Borse M.: “Comparitive study of brain tumor detection using morphological operations”, International Journal of Research in Engineering and Technology, 2015, 4, pp. 422–428. [24]. Vyavahare A.J., Thool R.C.: “Segmentation using region growing algorithm based on CLAHE for medical images”, Fourth International Conference on Advances in Recent Technologies in Communication and Computing, 2012, pp. 182–185. [25]. Tạp chí Nghiên cứu KH và CN Quân sự số 55, Tháng 6- 2018 ABSTRACT BRAIN TUMOR DETECTION AND SEGMENTATION USING EDGE AND REGION GROWING METHOD In the field of medical image processing, detection of brain tumor from computed tomography (CT) or magnetic resonance (MRI) scans is one of the top priority goals. In this article, we describe a new method which combines smoothing, Sobel edge detection, connected component and finally region growing algorithms for locating and extracting the various lesions automatically and efficiently. This combined activity will perform a cycle that includes the functions: test the brain image process, image filtering, segmentation, determination of the tumor location and extracts the tumor region. The computational algorithm was implemented using Insight Toolkit (ITK) to process input image, Visualization Toolkit (VTK) to display and Qt software development framework to build user interface. The analysis results indicate that the proposed method efficiently detected the tumor region from the brain image. It is very clear for physicians to separate the abnormal from the normal surrounding tissue to get a real identification of related and unrelated area for performing advanced image processing, improving quality and accuracy of diagnosis, increasing therapy success by early detection of tumor as well as reducing surgical planning time. Keywords: MRI image; CT image; Smoothing; Sobel; Region Growing; Tumor; Segmentation; ITK; VTK; Qt. Nhận bài ngày 10 tháng 7 năm 2018 Hoàn thiện ngày 27 tháng 7 năm 2018 Chấp nhận đăng ngày 10 tháng 8 năm 2018 Địa chỉ: 1Viện thiết bị và công trình Y tế, Hà Nội, Việt Nam ; 2Trung tâm Vật lý hạt nhân, Viện Vật lý, Viện Hàn lâm Khoa học và Công nghệ Việt Nam; 3Học viện Kỹ thuật quân sự. *Email: haquangthanh70@gmail.com.
File đính kèm:
 phat_hien_va_phan_tach_khoi_u_nao_su_dung_phuong_phap_dinh_b.pdf
phat_hien_va_phan_tach_khoi_u_nao_su_dung_phuong_phap_dinh_b.pdf

