Một phương pháp phân hạng gen gây bệnh mới dựa trên tổng xác suất liên kết trong mạng tương tác protein
Xác định các gen mới có liên quan đến bệnh là một bài toán quan trọng trong nghiên cứu y sinh. Đây có thể coi là bước khởi đầu trong việc tìm ra phương pháp điều trị cho các bệnh phát sinh do yếu tố di truyền

Trang 1

Trang 2

Trang 3

Trang 4

Trang 5
Trang 6
Trang 7
Trang 8
Trang 9

Trang 10
Tải về để xem bản đầy đủ
Bạn đang xem 10 trang mẫu của tài liệu "Một phương pháp phân hạng gen gây bệnh mới dựa trên tổng xác suất liên kết trong mạng tương tác protein", để tải tài liệu gốc về máy hãy click vào nút Download ở trên
Tóm tắt nội dung tài liệu: Một phương pháp phân hạng gen gây bệnh mới dựa trên tổng xác suất liên kết trong mạng tương tác protein

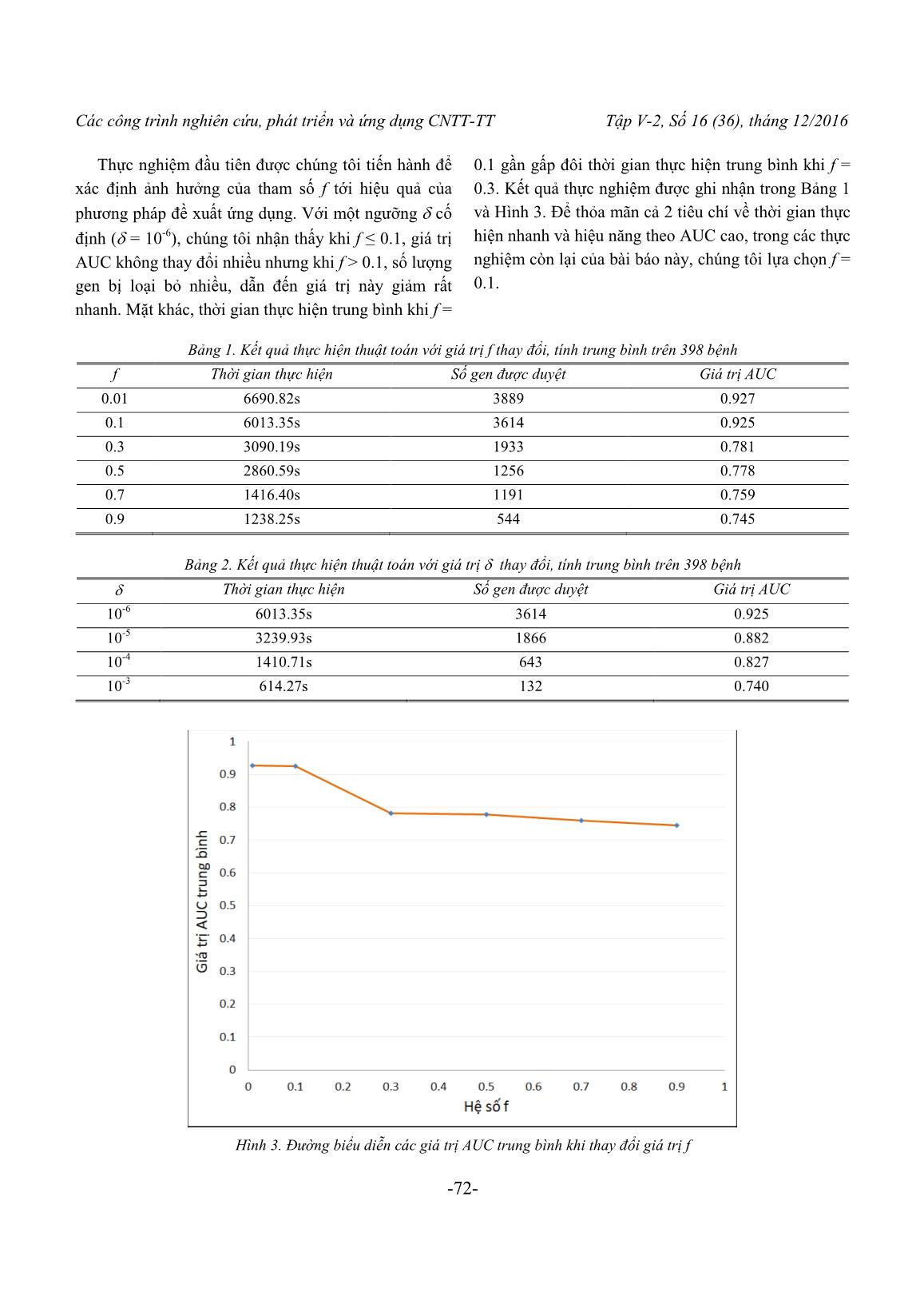
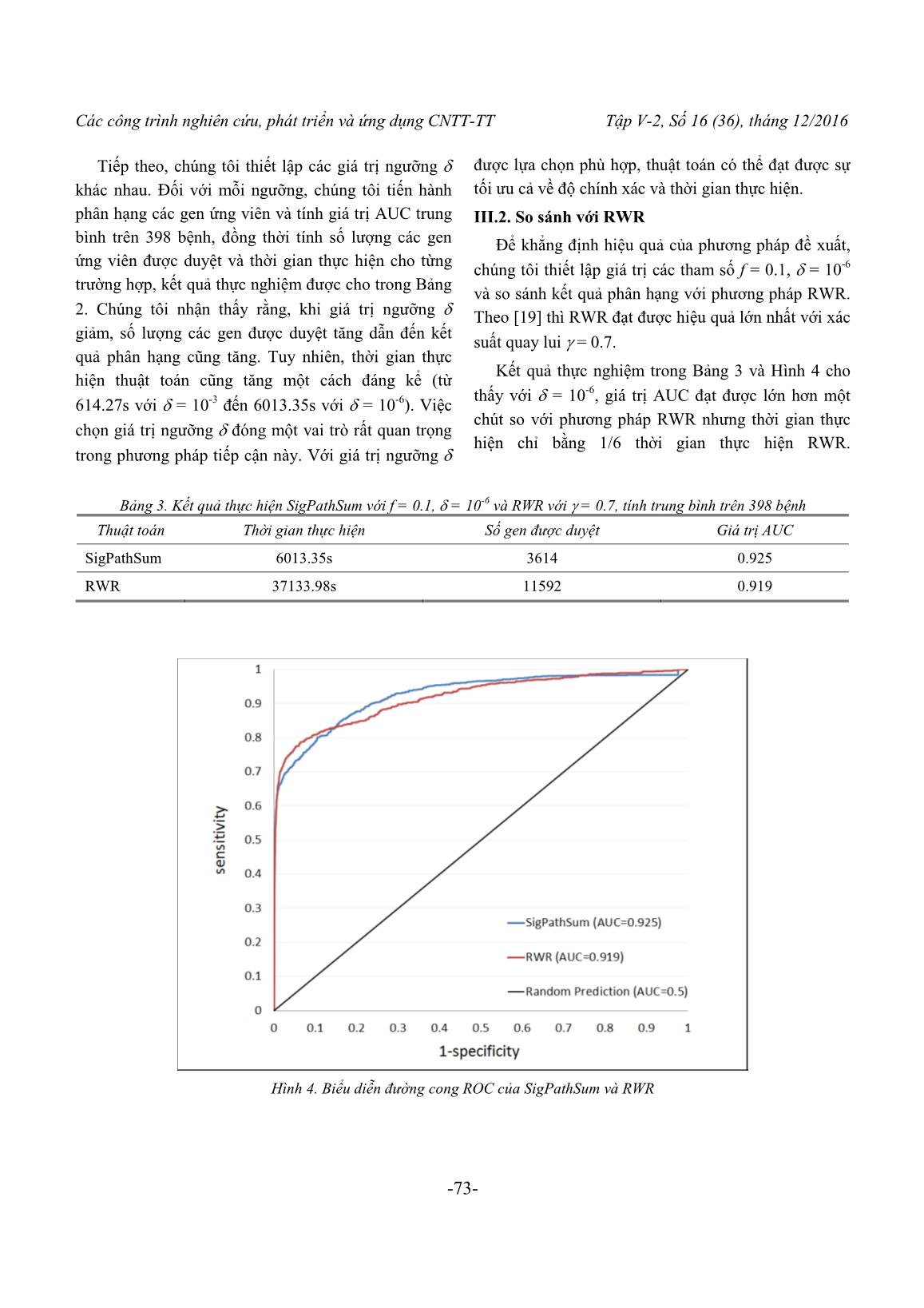
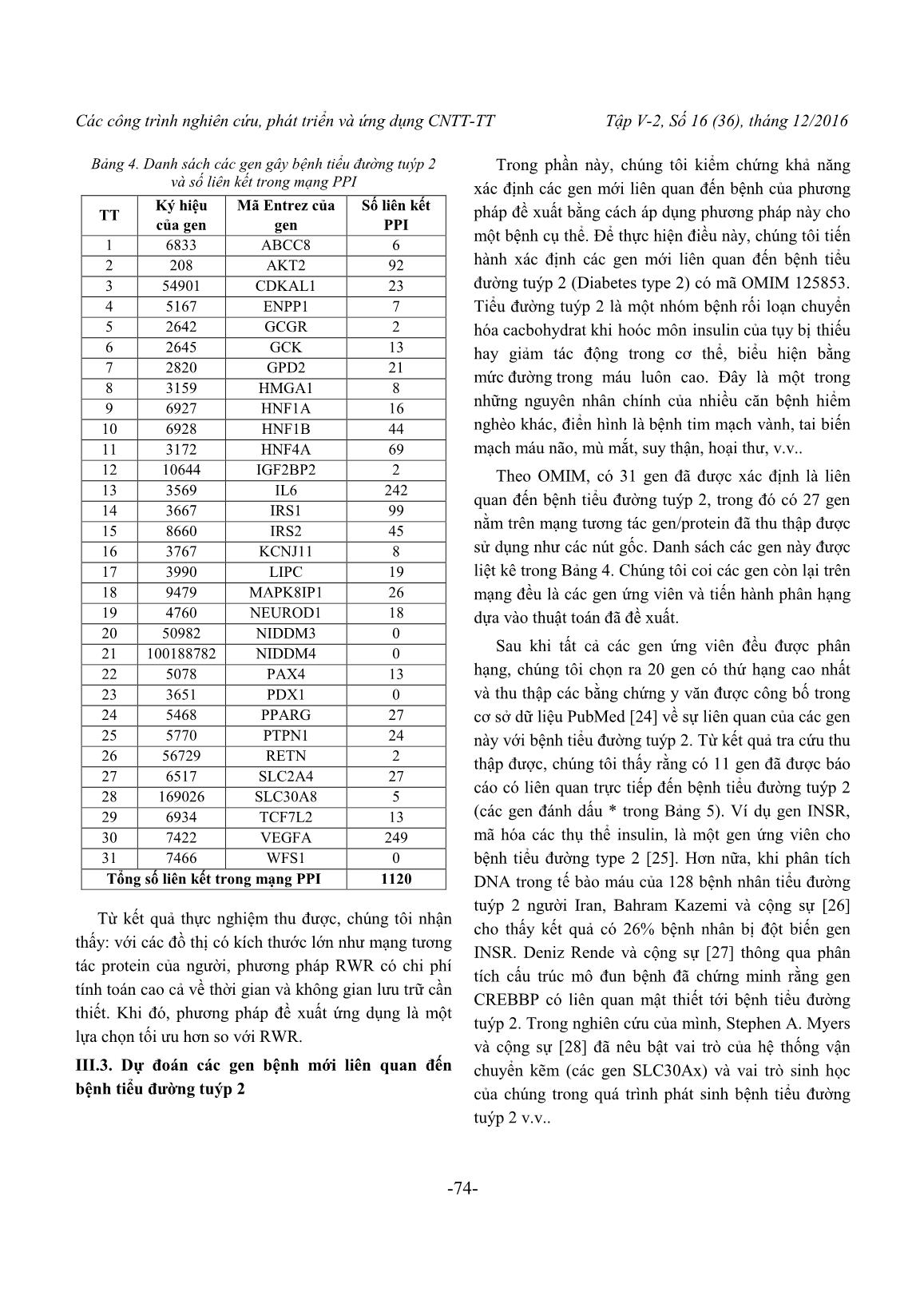
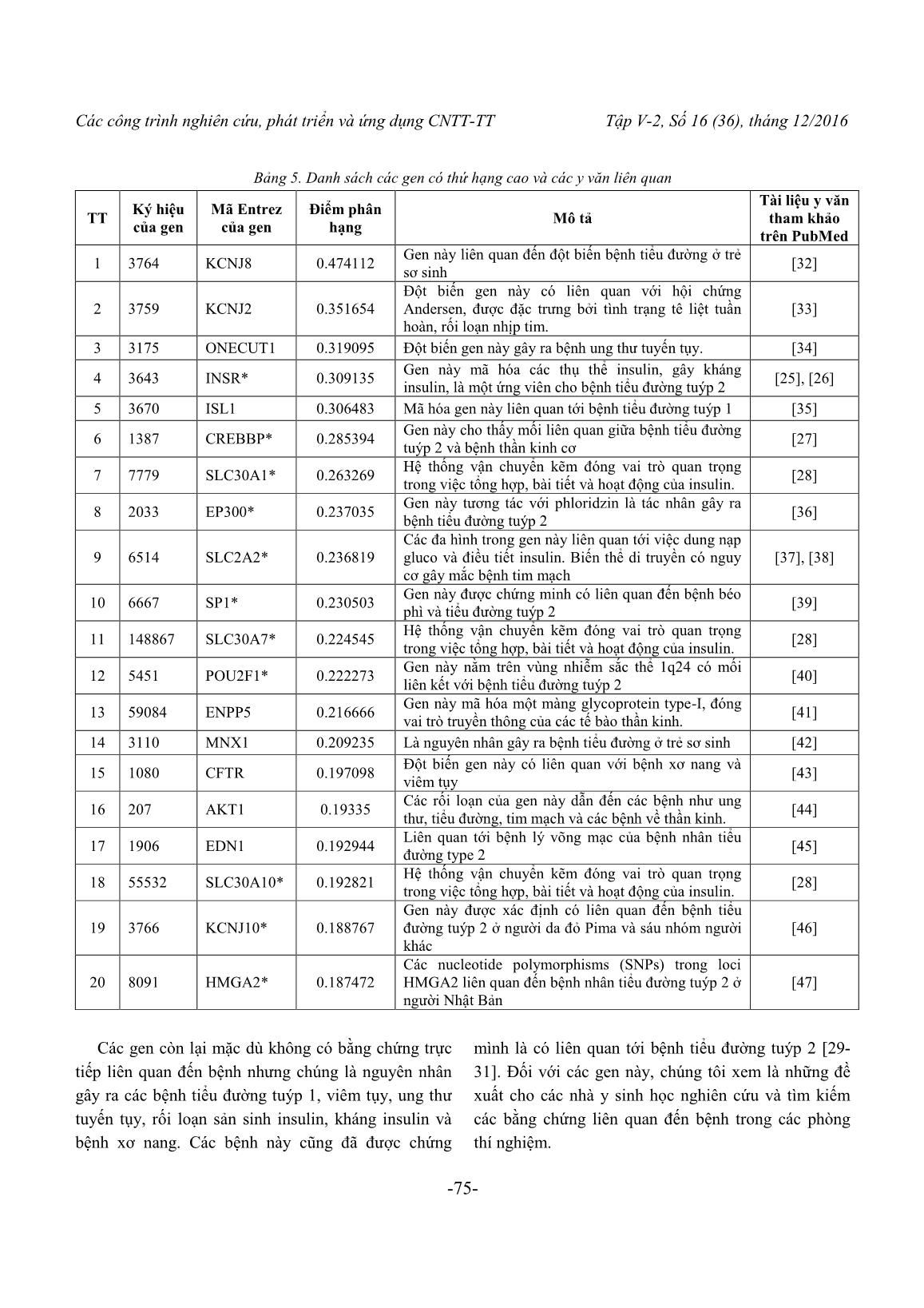
Các công trình nghiên cứu, phát triển và ứng dụng CNTT-TT Tập V-2, Số 16 (36), tháng 12/2016 -67- Một phƣơng pháp phân hạng gen gây bệnh mới dựa trên tổng xác suất liên kết trong mạng tƣơng tác protein A Novel Candidate Disease Genes Prioritization Method based on the Total Probability Links Protein Interaction Network Đặng Vũ Tùng, Nguyễn Đại Phong, Lê Đức Hậu, Từ Minh Phƣơng Abstract: Prioritizing candidate disease-related genes using computational methods and biological networks data is an important problem in bioinformatics. Random walk with restart (RWR) algorithm is widely used for this problem due to its relatively high accuracy. However, RWR is computationally expensive as it considers every node in a network. Here we propose to use a new method for prioritizing candidate genes, in which genes with low probability of association with disease genes are excluded from further consideration, thus reducing computational complexity. Experiments on real protein interaction networks show that the proposed method was computationally efficient, and more accurate than RWR, as measured by AUC scores. We applied the proposed method to prioritizing candidate genes for human diabetes type 2. The results were promising: among top 20 ranked genes, 11 are associated with diabetes, as reported in the biomedical literature. Keywords: Protein Interaction Network, Genes Prioritization, Type 2 Diabetes, RWR. I. MỞ ĐẦU Xác định các gen mới có liên quan đến bệnh là một bài toán quan trọng trong nghiên cứu y sinh. Đây có thể coi là bƣớc khởi đầu trong việc tìm ra phƣơng pháp điều trị cho các bệnh phát sinh do yếu tố di truyền [1-3]. Trong giai đoạn trƣớc đây, việc xác định gen gây bệnh đƣợc thực hiện chủ yếu bằng thực nghiệm sinh học để xác định các vùng nhiễm sắc thể khả nghi liên quan bệnh cần nghiên cứu [4, 5]. Tuy nhiên, những vùng nhiễm sắc thể này thƣờng chứa hàng trăm gen ứng viên, trong khi chỉ có một số ít các gen thực sự liên quan đến bệnh [6]. Để xác định đƣợc chính xác các gen liên quan đến bệnh cần nghiên cứu, các nhà y sinh học phải tiến hành các thí nghiệm cho từng gen trong danh sách gen ứng viên thu đƣợc. Đây là công việc rất tốn kém về thời gian và kinh phí. Các khó khăn này hiện nay đã đƣợc giải quyết một phần bằng phƣơng pháp phân hạng gen ứng viên liên quan đến bệnh trong Tin sinh học. Mục đích của việc phân hạng các gen ứng viên theo mức độ liên quan đến một căn bệnh là để xác định các gen mới có liên quan đến bệnh. Cho đến nay, đã có nhiều phƣơng pháp tính toán đƣợc phát triển nhằm mục đích phân hạng các gen ứng viên liên quan đến các bệnh di truyền [7-11]. Trong giai đoạn đầu, các phƣơng pháp tính toán chủ yếu dựa trên dữ liệu chú giải chức năng. Theo đó, mức độ liên quan của gen ứng viên và bệnh nghiên cứu căn cứ vào độ tƣơng tự về hồ sơ chức năng đƣợc xây dựng từ các dữ liệu chú giải của gen ứng viên và các gen bệnh đã biết [7, 9, 10]. Tuy nhiên, hạn chế của các phƣơng pháp này đó là các dữ liệu chú giải chức năng thƣờng không đầy đủ cho tất cả các gen/protein. Điều này ảnh hƣởng đến việc xây dựng các hồ sơ chức năng cho tất cả các gen. Gần đây, các phƣơng pháp tính toán đƣợc chuyển theo hƣớng dựa trên các mạng sinh học do dữ liệu về tƣơng tác giữa các gen/protein ngày càng đầy đủ và có thể bao phủ toàn bộ hệ gen. Các phƣơng pháp này thƣờng căn cứ vào nguyên lý “mô đun bệnh” (nghĩa là, các gen/protein liên quan đến cùng một bệnh hoặc các Các công trình nghiên cứu, phát triển và ứng dụng CNTT-TT Tập V-2, Số 16 (36), tháng 12/2016 -68- bệnh tƣơng tự nhau có xu hƣớng nằm kề nhau trong các mạng tƣơng tác [4]) để tính toán độ tƣơng tự tƣơng giữa các gen ứng viên và các gen gây bệnh đã biết. Có rất nhiều phƣơng pháp dựa trên mạng đã đƣợc đề xuất cho bài toán này nhƣ: dựa trên các láng giềng gần nhất, dựa trên các cụm trên mạng. Ngoài ra, các thuật toán ph biến trong phân tích mạng xã hội và mạng Web dùng để đánh giá tầm quan trọng tƣơng đối của nút nhƣ: HITS with priors, PageRank with priors, K-step Markov [12], RL_Rank [13] và bƣớc ngẫu nhiên có quay lui (RWR) [14] cũng đã đƣợc sử dụng cho bài toán phân hạng các gen ứng viên trên các mạng tƣơng tác protein. Trong số đó, phƣơng pháp RWR đƣợc đánh giá là phƣơng pháp n i trội nhất [15]. Phƣơng pháp này khai thác cấu trúc t ng thể của mạng dựa vào hành vi của một chuyển động ngẫu nhiên trên một mạng hay đồ thị. Theo hành vi này, một thực thể xuất phát từ một nút khởi đầu sau đó di chuyển trên đồ thị bằng cách chuyển đến các nút lân cận một cách ngẫu nhiên với xác suất tỷ lệ với trọng số của các cạnh kết nối. Tại thời điểm bất kỳ trong quá trình di chuyển, thực thể cũng có thể quay lại nút khởi đầu với một xác suất nhất định đƣợc gọi là xác suất quay lui (back- probability). Các nút trên đồ thị đƣợc thăm nhiều hơn sẽ đƣợc xem là có độ quan trọng lớn hơn, đại lƣợng này đánh giá tầm quan trọng tƣơng đối/độ liên quan của các nút còn lại so với tập các nút gốc. Khi áp dụng thuật toán này cho bài toán phân hạng gen gây bệnh, các gen gây bệnh đã biết đóng vai trò nhƣ các nút khởi đầu, các gen còn lại trên mạng đƣợc xem là các ứng viên. Kohler và cộng sự [14] đã áp dụng thuật toán này trên các mạng tƣơng tác protein để xác định các gen gây bệnh mới. Kết quả thử nghiệm trên một tập gồm 110 bệnh cho thấy phƣơng pháp này đạt đƣợc hiệu năng dự đoán tốt và cao hơn so với các phƣơng pháp dựa trên dữ liệu chú giải chức năng. Không những đạt đƣợc hiệu năng cao trong bài toán phân hạng gen ứng viên liên quan đến bệnh, thuật toán này còn đƣợc sử dụng hiệu quả trong việc các định các microRNA mới liên quan đến bệnh [16] cũng nhƣ các đích tác động mới của thuốc [17]. Tuy nhiên, RWR phải duyệt qua tất cả các nút trên đồ thị thông qua các phép nhân ma trận, do đó nó có độ phức tạp tính toán cao đối với các đồ thị lớn nhƣ các mạng sinh ... g chứng liên quan đến bệnh trong các phòng thí nghiệm. Các công trình nghiên cứu, phát triển và ứng dụng CNTT-TT Tập V-2, Số 16 (36), tháng 12/2016 -76- IV. KẾT LUẬN Trong bài báo này, chúng tôi đã đề xuất ứng dụng một thuật toán mới trong phân tích mạng xã hội, mạng web để phân hạng và tìm kiếm các gen ứng viên có độ liên quan cao nhất đối với các gen bệnh đã biết dựa trên t ng xác suất đƣờng đi giữa hai gen/protein trong mạng. Thực nghiệm cho thấy khi sử dụng một giá trị ngƣỡng nhất định ( = 10-6) kết quả phân hạng đạt đƣợc tốt hơn so với phƣơng pháp dựa trên thuật toán RWR nhƣng với thời gian thực hiện ít hơn. Chú ý rằng, mạng tƣơng tác gen/protein có thể đƣợc hình thành bởi các tƣơng tác vật lý giữa chúng hoặc có thể đƣợc xây dựng dựa trên độ tƣơng tự về chức năng giữa các gen/protein trên mạng. Dẫn đến, các mạng gen/protein có thể có kích thƣớc rất lớn để phản ánh đầy đủ mối quan hệ chức năng phức tạp giữa các thành phần trong tế bào. Phƣơng pháp này đƣợc đề xuất để áp dụng trên các mạng tƣơng tác gen/protein có kích thƣớc lớn trong khi vẫn đảm bảo hiệu năng dự đoán cao. Kết quả thực nghiệm cũng cho thấy ngoài đạt đƣợc hiệu năng t ng thể cao, phƣơng pháp này còn có thể sử dụng để xác định các gen mới liên quan đến một bệnh cụ thể. Các gen có thứ hạng cao nhƣng chƣa có bằng chứng y sinh trực tiếp về mối liên quan giữa chúng với bệnh xem xét có thể đƣợc đề xuất để các nhà nghiên cứu y sinh học tiếp tục nghiên cứu thực nghiệm. Với các kết quả nghiên cứu và thực nghiệm đã thu đƣợc, chúng tôi hy vọng có thể phát triển phƣơng pháp đề xuất ứng dụng thành công cụ tìm kiếm gen gây bệnh trong tƣơng lai nhƣ [48]. Thêm vào đó, với sự gia tăng không ngừng của các dữ liệu sinh học, nhiều mạng sinh học cũng đƣơc cấu thành dựa trên các dữ liệu này. Việc tích hợp nhiều loại dữ liệu liên quan đến bệnh sẽ cải thiện hiệu năng của các thuật toán dựa trên mạng, cũng nhƣ tạo động lực để đề xuất các thuật toán mới hiệu quả hơn [19]. Thật vậy, bằng việc tích hợp thêm dữ liệu về độ tƣơng tự giữa các kiểu hình bệnh, Li và cộng sự [49] đã sử dụng thuật toán bƣớc ngẫu nhiên có khởi động lại cho mạng không đồng nhất bằng cách kết hợp mạng gen và mạng kiểu hình. LỜI CẢM ƠN Nghiên cứu này đƣợc tài trợ bởi Quỹ phát triển khoa học và công nghệ quốc gia (NAFOSTED) trong đề tài mã số 102.01-2014.21. TÀI LIỆU THAM KHẢO [1] G. H. FERNALD, E. CAPRIOTTI, R. DANESHJOU, K. J. KARCZEWSKI and R. B. ALTMAN, "Bioinformatics challenges for personalized medicine", Bioinformatics, 27 (2011), pp. 1741-1748. [2] D. JONES, "Steps on the road to personalized medicine", Nature Reviews Drug Discovery, 6 (2007), pp. 770-771. [3] K. REYNOLDS, "Achieving the Promise of Personalized Medicine", Clinical Pharmacology & Therapeutics, 92 (2012), pp. 401-405. [4] S. R, U. I and S. R, "Network-based prediction of protein function", Molecular Systems Biology, 3(88) (2007). [5] M. ML, M. JC, L. AC, A.-B. M, C. ME and E. AL, "Meta-analysis of 13 genome scans reveals multiple cleft lip/palate genes with novel loci on 9q21 and 2q32-35", American Journal of Human Genetics, 75(2) (2004), pp. 161-173. [6] J. LB, "Linkage disequilibrium and the search for complex disease genes", Genome Research, 10(10) (2000), pp. 1435-1444. [7] A. EA, A. RR, E. KL, P. DJ and P. BS, "Suspects: enabling fast and effective prioritization of positional candidates", Bioinformatics, 22 (2006), pp. 773-774. [8] H. JE, K. AT, M. HL and P. MA, "Candid: a flexible method for prioritizing candidate genes for complex human traits", Genetic Epidemiology, 32 (2008), pp. 779-790. [9] A. S, L. D, M. S, V. L. P, C. B and E. AL, "Gene prioritization through genomic data fusion", Nature Biotechnology, 24 (2006), pp. 537-544. [10] C. J, X. H, A. BJ and J. AG, "Improved human disease candidate gene prioritization using mouse phenotype", BMC Bioinformatics, 8 (2007). [11] S. D, S. JM and S. M, "Genedistiller - distilling candidate genes from linkage intervals", PLoS ONE,, 3 (2008). Các công trình nghiên cứu, phát triển và ứng dụng CNTT-TT Tập V-2, Số 16 (36), tháng 12/2016 -77- [12] C. J., A. B. and J. A., "Disease candidate gene identification and prioritization using protein interaction networks", BMC Bioinformatics, 10 (2009). [13] Đ. V. TÙNG, D. A. TRÀ, L. Đ. HẬU and T. M. PHƢƠNG, "Phân hạng gen gây bệnh sử dụng học tăng cường kết hợp với xác suất tiền nghiệm", Tạp chí Công nghệ thông tin & Truyền thông, 13(33) (2015), pp. 55-66. [14] S. KÖHLER, S. BAUER, D. HORN and P. N. ROBINSON, "Walking the Interactome for Prioritization of Candidate Disease Genes", The American Journal of Human Genetics, 82 (2008), pp. 949-958. [15] S. NAVLAKHA and C. KINGSFORD, "The power of protein interaction networks for associating genes with diseases.", Bioinformatics 26 (2010), pp. 1057-1063. [16] D.-H. LE, "Network-based ranking methods for prediction of novel disease associated microRNAs", Computational Biology and Chemistry, 58 (2015), pp. 139-148. [17] X. CHEN, M.-X. LIU and G.-Y. YAN, "Drug– target interaction prediction by random walk on the heterogeneous network", Molecular BioSystems, 8 (2012), pp. 1970-1978. [18] H. WANG, C. K. CHANG, H.-I. YANG and Y. CHEN, "Estimating the Relative Importance of Nodes in Social Networks", Journal of Information Processing Society of Japan, 21(3) (2013), pp. 414- 422. [19] D.-H. LE and Y.-K. KWON, "Neighbor-favoring weight reinforcement to improve random walk- based disease gene prioritization", Computational Biology and Chemistry, 44 (2013), pp. 1-8. [20] B. LINGHU, E. S. SNITKIN, Z. HU, Y. XIA and C. DELISI, "Genome-wide prioritization of disease genes and identification of disease-disease associations from an integrated human functional linkage network", Genome Biology, 10 (2009). [21] J. AMBERGER, C. A. BOCCHINI, A. F. SCOTT and A. HAMOSH, "McKusick's Online Mendelian Inheritance in Man (OMIM®)", Nucleic Acids Research, 37 (2009), pp. D793-D796. [22] D. J. WATTS and S. H. STROGATZ, "Collective dynamics of small-world networks", Nature 393(1) (1998), pp. 440-442. [23] B. H. JUNKER, D. KOSCHÜTZKI and F. SCHREIBER, "Exploration of biological network centralities with CentiBiN", BMC Bioinformatics, 7:219 (2006). [24] J. D. OSBORNE, S. LIN, W. A. KIBBE, L. J. ZHU, M. I. DANILA and R. L. CHISHOLM, "GeneRIF is a more comprehensive, current and computationally tractable source of gene-disease relationships than OMIM", Oxford University Press (2006). [25] B. D, S. M, G. S, M. PP, R. MR, M. V and R. V, "Association of His1085His INSR gene polymorphism with type 2 diabetes in South Indians", Diabetes Technol Ther, 14 (2012), pp. 696-700. [26] B. KAZEMI, N. SEYED, E. MOSLEMI, M. BANDEHPOUR, M. B. TORBATI, N. SAADAT, A. EIDI, E. GHAYOOR and F. AZIZI, "Insulin Receptor Gene Mutations in Iranian Patients with Type II Diabetes Mellitus", Iranian Biomedical Journal, 13 (2009), pp. 161-168. [27] D. RENDE, N. BAYSAL and B. KIRDAR, "Complex Disease Interventions from a Network Model for Type 2 Diabetes", PLoS One, 8 (2013). [28] S. A. MYERS, A. NIELD and M. MYERS, "Zinc Transporters, Mechanisms of Action and Therapeutic Utility: Implications for Type 2 Diabetes Mellitus", Journal of Nutrition and Metabolism, 2012 (2012), pp. 13. [29] C. S. C. RICHARD I. G. HOLT, ALLAN FLYVBJERG, BARRY J. GOLDSTEIN, Textbook of Diabetes, Wiley-Blackwell, 2010. [30] L. PORETSKY, Principles of Diabetes Mellitus, Springer New York Dordrecht Heidelberg London, 2010. [31] R. TAYLOR, "Insulin Resistance and Type 2 Diabetes", Diabetes, 61 (2012), pp. 778-779. [32] M. WINKLER, R. LUTZ, U. RUSS, U. QUAST and J. BRYAN, "Analysis of two KCNJ11 neonatal diabetes mutations, V59G and V59A, and the analogous KCNJ8 I60G substitution: differences between the channel subtypes formed with SUR1.", J Biol Chem, 284 (2009), pp. 6752-6762. [33] K.-P. A, P.-C. A, P. P, B. K, M.-K. M, B. P, S. K, L. HY, Q. E, P. R, K. A and P. LJ, "Andersen-Tawil syndrome: report of 3 novel mutations and high risk of symptomatic cardiac involvement", Muscle Nerve, 51 (2015), pp. 192-196. Các công trình nghiên cứu, phát triển và ứng dụng CNTT-TT Tập V-2, Số 16 (36), tháng 12/2016 -78- [34] X. JIANG, W. ZHANG, H. KAYED, P. ZHENG, N. A. GIESE, H. FRIESS and J. KLEEFF, "Loss of ONECUT1 expression in human pancreatic cancer cells", Oncol Rep, 19 (2008), pp. 157-163. [35] P. HOLM, B. RYDLANDER, H. LUTHMAN and I. KOCKUM, "Interaction and Association Analysis of a Type 1 Diabetes Susceptibility Locus on Chromosome 5q11-q13 and the 7q32 Chromosomal Region in Scandinavian Families", Diabetes, 53 (2004), pp. 1584-1591. [36] V. RANDHAWA, P. SHARMA, S. BHUSHAN and G. BAGLER, "Identification of Key Nodes of Type 2 Diabetes Mellitus Protein Interactome and Study of their Interactions with Phloridzin", OMICS: A Journal of Integrative Biology, 17 (2013), pp. 302-317. [37] A. BORGLYKKE, N. GRARUP, T. SPARSØ, A. LINNEBERG, M. FENGER, J. JEPPESEN, T. HANSEN, O. PEDERSEN and T. JØRGENSEN, "Genetic Variant SCL2A2 Is Associated with Risk of Cardiovascular Disease – Assessing the Individual and Cumulative Effect of 46 Type 2 Diabetes Related Genetic Variants", PLoS One, 7 (2012). [38] O. LAUKKANEN, J. LINDSTRÖM, J. ERIKSSON, T. T. VALLE, H. HÄMÄLÄINEN, P. ILANNE-PARIKKA, S. KEINÄNEN- KIUKAANNIEMI, J. TUOMILEHTO, M. UUSITUPA and M. LAAKSO, "Polymorphisms in the SLC2A2 (GLUT2) Gene Are Associated With the Conversion From Impaired Glucose Tolerance to Type 2 Diabetes: The Finnish Diabetes Prevention Study", Diabetes, 54 (2005), pp. 2256-2260. [39] J. CHEN, Y. MENG, J. ZHOU, M. ZHUO, F. LING, Y. ZHANG, H. DU and X. WANG, "Identifying Candidate Genes for Type 2 Diabetes Mellitus and Obesity through Gene Expression Profiling in Multiple Tissues or Cells", J Diabetes Res, 2013 (2013). [40] N. MC, L. VK, T. CH, C. AW, S. WY, M. RC, Z. BC, W. MM, M. WW, H. C, W. CR, T. PC, J. WP and C. JC, "Association of the POU class 2 homeobox 1 gene (POU2F1) with susceptibility to Type 2 diabetes in Chinese populations", Diabetic Medicine, 27 (2010), pp. 1443-1449. [41] REFSEQ, ENPP5 ectonucleotide pyrophosphatase/phosphodiesterase 5, 2014. [42] B. A, V. E, P. J, S. B, L. S, Y. L, H. M, C. H, B. K, S. R, P. M, A.-R. M, F. P and V. M, "Transcription factor gene MNX1 is a novel cause of permanent neonatal diabetes in a consanguineous family", Diabetes Metab, 39 (2013), pp. 276-280. [43] S. KONDO, K. FUJIKI, S. B. H. KO, A. YAMAMOTO, M. NAKAKUKI, Y. ITO, N. SHCHEYNIKOV, M. KITAGAWA, S. NARUSE and H. ISHIGURO, "Functional characteristics of L1156F-CFTR associated with alcoholic chronic pancreatitis in Japanese", American Journal of Physiology - Gastrointestinal and Liver Physiology, 309 (2015), pp. 260-269. [44] I. HERSA, E. E. VINCENT and J. M. TAVARÉ, "Akt signalling in health and disease", Cellular Signalling, 23 (2011), pp. 1515-1527. [45] H. LI, J. W. C. LOUEY, K. W. CHOY, D. T. L. LIU, W. M. CHAN, Y. M. CHAN, N. S. K. FUNG, B. J. FAN, L. BAUM, J. C. N. CHAN, D. S. C. LAM and C. P. PANG, "EDN1 Lys198Asn is associated with diabetic retinopathy in type 2 diabetes", Molecular Vision, 2008 (2008), pp. 1698- 1704. [46] V. S. FAROOK, R. L. HANSON, J. K. WOLFORD, C. BOGARDUS and M. PROCHAZKA, "Molecular Analysis of KCNJ10 on 1q as a Candidate Gene for Type 2 Diabetes in Pima Indians", Diabetes, 51 (2002), pp. 3342-3346. [47] T. OHSHIGE, M. IWATA, S. OMORI, Y. TANAKA, H. HIROSE, K. KAKU, H. MAEGAWA, H. WATADA, A. KASHIWAGI, R. KAWAMORI, K. TOBE, T. KADOWAKI, Y. NAKAMURA and S. MAEDA, "Association of New Loci Identified in European Genome-Wide Association Studies with Susceptibility to Type 2 Diabetes in the Japanese", PLoS One, 6 (2011). [48] D.-H. LE and Y.-K. KWON, "GPEC: A Cytoscape plug-in for random walk-based gene prioritization and biomedical evidence collection", Computational Biology and Chemistry, 37 (2012), pp. 17-23. [49] L. Y and P. JC, "Genome-wide inferring gene- phenotype relationship by walking on the heterogeneous network", Bioinformatics, 26 (2010), pp. 1219-1224. Nhận bài ngày: 13/03/2016 Các công trình nghiên cứu, phát triển và ứng dụng CNTT-TT Tập V-2, Số 16 (36), tháng 12/2016 -79- SƠ LƢỢC VỀ TÁC GIẢ ĐẶNG VŨ TÙNG Sinh năm 1972. Tốt nghiệp ĐH T ng hợp Hà Nội năm 1995; Thạc sỹ chuyên ngành Hệ thống thông tin năm 2011; NCS khóa 2013 tại Học viện Công nghệ bƣu chính viễn thông. Hiện công tác tại bộ môn Tin học, Học viện Thanh thiếu niên Việt Nam. Lĩnh vực nghiên cứu: hệ thống thông tin, tin sinh học. Điện thoại: 0913542479 Email: tung_dv@yahoo.com NGUYỄN ĐẠI PHONG Sinh năm 1993. Sinh viên trƣờng ĐH Bách Khoa Hà Nội. Lĩnh vực nghiên cứu: lập trình Matlab, tin sinh học. Điện thoại: 0973794518 Email: phongnd.hust@gmail.com LÊ ĐỨC HẬU Sinh năm 1979. Tốt nghiệp ĐH Bách khoa Hà Nội năm 2002; Thạc sỹ khoa học ĐH Bách Khoa Hà nội năm 2008; Bảo vệ Tiến sĩ năm 2012 tại ĐH Ulsan, Hàn Quốc. Hiện công tác tại Trung tâm Tin học, ĐH Thủy Lợi. Lĩnh vực nghiên cứu: học máy và khai phá dữ liệu, tin sinh học và ứng dụng, phân tích và khai phá mạng xã hội, lập trình song song trên GPU với CUDA và OpenCL. Điện thoại: 0912324564 Email: hauldhut@gmail.com TỪ MINH PHƢƠNG Sinh năm 1971. Tốt nghiệp ĐH Bách khoa Taskent, Uzobekistan; Bảo vệ tiến sĩ năm 1995 tại Viện Điều khiển học thuộc Viện hàn lâm khoa học Uzobekistan. Hiện công tác tại Khoa CNTT, Học viện Công nghệ Bƣu chính viễn thông. Lĩnh vực nghiên cứu: ứng dụng của học máy, tin sinh học. Điện thoại: 0913507508 Email: phuongtm@ptit.edu.vn
File đính kèm:
 mot_phuong_phap_phan_hang_gen_gay_benh_moi_dua_tren_tong_xac.pdf
mot_phuong_phap_phan_hang_gen_gay_benh_moi_dua_tren_tong_xac.pdf

